WGS Workflow: Grotere weergave
Download pdf-pictogram
Wat is whole genome sequencing (WGS)?
Het genoom, of genetisch materiaal, van een organisme (bacterie, virus, aardappel, mens) is opgebouwd uit DNA. Elk organisme heeft een unieke DNA-sequentie die is opgebouwd uit basen (A, T, C, en G). Als je de volgorde van de basen in een organisme kent, heb je zijn unieke DNA-vingerafdruk, of patroon, geïdentificeerd. Het bepalen van de volgorde van de basen wordt sequencing genoemd. Whole genome sequencing is een laboratoriumprocedure waarmee de volgorde van de basen in het genoom van een organisme in één proces wordt bepaald.
Hoe werkt whole genome sequencing?
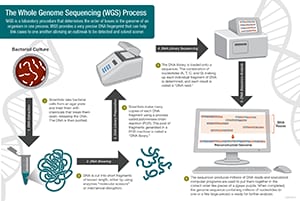
Wetenschappers voeren whole genome sequencing uit door deze vier hoofdstappen te volgen:
- DNA afschuiven: Wetenschappers beginnen met een moleculaire schaar om het DNA, dat uit miljoenen basen bestaat, door te knippen: A’s, C’s, T’s en G’s, in stukjes die klein genoeg zijn voor de sequencing-machine om te lezen.
- DNA-barcoding: Wetenschappers voegen kleine stukjes DNA-tags, of streepjescodes, toe om te identificeren welk stukje geschoren DNA bij welke bacterie hoort. Dit is vergelijkbaar met de manier waarop een streepjescode een product in een supermarkt identificeert.
- Volkomen genoom sequencing: Het DNA met streepjescode van meerdere bacteriën wordt gecombineerd en in de whole genome sequencer gestopt. De sequencer identificeert de A’s, C’s, T’s, en G’s, of basen, die deel uitmaken van elke bacteriële sequentie. De sequencer gebruikt de streepjescode om bij te houden welke basen bij welke bacteriën horen.
- Gegevensanalyse: Wetenschappers gebruiken computeranalyse-instrumenten om bacteriële sequenties te vergelijken en verschillen te identificeren. Het aantal verschillen kan de wetenschappers vertellen hoe nauw verwant de bacteriën zijn, en hoe waarschijnlijk het is dat ze deel uitmaken van dezelfde uitbraak.

Hoe zal whole genome sequencing het opsporen van ziekten transformeren?
Whole genome sequencing levert gedetailleerdere en preciezere gegevens op voor het identificeren van uitbraken dan de huidige standaardtechniek die PulseNet gebruikt, pulsed-field gel electrophoresis (PFGE). In plaats van alleen de mogelijkheid te hebben om bacteriële genomen te vergelijken aan de hand van 15-30 banden die in een PFGE-patroon verschijnen, hebben we nu miljoenen basen om te vergelijken. Dat is alsof je alle woorden in een boek vergelijkt (WGS), in plaats van alleen het aantal hoofdstukken (PFGE), om te zien of de boeken hetzelfde of verschillend zijn. Met behulp van whole genome sequencing hebben wij ontdekt dat sommige bacteriën die met PFGE verschillend leken, in feite van dezelfde bron afkomstig zijn. Dit heeft ertoe bijgedragen dat sommige uitbraken sneller konden worden opgelost.
Whole genome sequencing is een snelle en betaalbare manier om met slechts één test informatie van hoog niveau over de bacterie te verkrijgen. Momenteel vereist het proces om bacteriën volledig te karakteriseren twee of meer wetenschappers om vier of meer afzonderlijke tests uit te voeren, waaronder PFGE. WGS zal de efficiëntie van hoe PulseNet surveillance uitvoert sterk verbeteren.
PulseNet is actief bezig met het valideren van next-generation sequencing (NGS) technologie, evenals het ontwikkelen, evalueren en implementeren van de tools die nodig zijn om de gegevens te analyseren.
Het hoofd bieden aan de uitdagingen van whole genome sequencing.
In 2013 begon CDC whole genome sequencing te gebruiken om uitbraken veroorzaakt door de dodelijke bacterie Listeria te detecteren. Sindsdien heeft deze methode wetenschappers in staat gesteld om:
- Meer clusters van Listeria-ziekten opsporen
- Meer Listeria-uitbraken oplossen terwijl ze nog klein zijn
- Zieke patiënten koppelen aan waarschijnlijke voedselbronnen
- Nieuwe voedselbronnen van Listeria identificeren, zoals karamel appels en ijs
Lees meer over hoe het Listeria Whole Genome Sequencing Project de opsporing en het onderzoek van door voedsel overgedragen uitbraken heeft verbeterd.
Het CDC breidt het gebruik van whole genome sequencing in staatslaboratoria snel uit, en wetenschappers zullen binnenkort beginnen met het gebruik van whole genome sequencing voor uitbraakonderzoeken van andere ziekteverwekkers in voedsel, zoals Campylobacter, Shiga toxine-producerende E. coli (STEC), en Salmonella. CDC’s Advanced Molecular Detection (AMD) initiatief financierde gedeeltelijk de uitbreiding van real-time WGS voor voedselveiligheid.
Door samenwerking met CDC’s AMD initiatief en het voedselveiligheidsprogramma, zet PulseNet de structuur op om een omschakeling naar whole genome sequencing te ondersteunen, inclusief het opleiden van volksgezondheidsmicrobiologen om sequencing uit te voeren, het aanschaffen van sequencing benodigdheden, en het updaten van systemen voor data-analyse. Deze activiteiten zijn van cruciaal belang om whole genome sequencing in volksgezondheidslaboratoria te introduceren en het toezicht op uitbraken van door voedsel overgedragen ziekten en trends in door voedsel overgedragen infecties en antibioticaresistentie te verbeteren.
Naarmate het gebruik van whole genome sequencing toeneemt, moeten de nationale surveillancesystemen en de laboratoriuminfrastructuur van CDC gelijke tred houden met de veranderende technologie. Door modernisering kunnen CDC en zijn partners op het gebied van de volksgezondheid infectieziekten met succes blijven opsporen, bestrijden en stoppen. Samen kunnen we zorgen voor snelle en minder dure diagnoses voor individuen en voor het bewijsmateriaal dat nodig is om uitbraken van door voedsel overgedragen ziekten snel op te lossen en te voorkomen.