Spektrometria mas jest techniką analityczną, w której próbki są jonizowane do naładowanych cząsteczek i stosunek ich mas do ładunków (m/z) może być mierzony. W spektrometrii mas MALDI-TOF, źródłem jonów jest wspomagana matrycą desorpcja/jonizacja laserowa (MALDI), a analizatorem mas jest analizator czasu przelotu (TOF).
MALDI
MALDI jest miękką jonizacją, która polega na uderzeniu lasera w matrycę małych cząsteczek, aby cząsteczki analitu przeszły do fazy gazowej bez ich fragmentacji lub rozkładu. Niektóre biomolekuły są zbyt duże i mogą ulec rozkładowi podczas ogrzewania, a tradycyjne techniki spowodują fragmentację lub zniszczenie makromolekuł. MALDI jest odpowiednia do analizy biomolekuł takich jak peptydy, lipidy, sacharydy i inne organiczne makrocząsteczki.
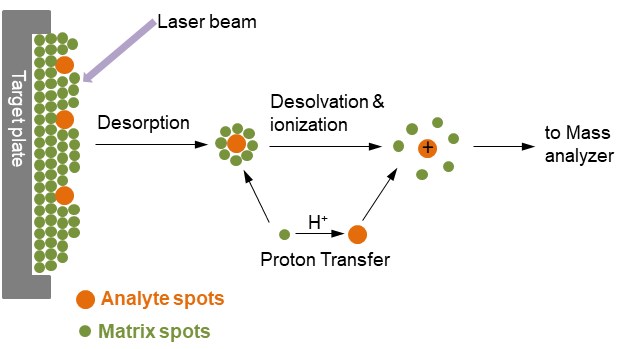
Rysunek 1. Jonizacja analitów metodą MALDI
- Zasada działania MALDI
Na rysunku 1, analit jest osadzony w bardzo dużym nadmiarze związku matrycowego osadzonego na stałej powierzchni zwanej targetem, zwykle wykonanej z metalu przewodzącego i posiadającej miejsca na kilka różnych próbek do naniesienia. Po bardzo krótkim impulsie laserowym, napromieniowana plamka jest gwałtownie podgrzewana i ulega wibracyjnemu wzbudzeniu. Cząsteczki matrycy energicznie ablowane z powierzchni próbki, absorbują energię lasera i przenoszą cząsteczki analitu również do fazy gazowej. Podczas procesu ablacji, cząsteczki analitu są zwykle jonizowane poprzez protonowanie lub deprotonowanie z pobliskimi cząsteczkami matrycy. Najbardziej powszechnym formatem jonizacji w MALDI jest to, że cząsteczki analitu posiadają pojedynczy ładunek dodatni.
- Typy laserów powszechnie stosowanych w MALDI
Wykorzystywane są lasery zarówno o długości fali ultrafioletowej (UV) jak i podczerwonej (IR), ale lasery UV są zdecydowanie najważniejszym źródłem światła w analitycznym MALDI. Spośród nich, lasery azotowe oraz potrójne lub poczwórne lasery Nd: Yag często służą do większości zastosowań. IR-MALDI jest zdominowane przez lasery Er:Yag, podczas gdy lasery TEA-CO2 są rzadko używane.
- Często używana substancja matrycy MALDI
Uważa się, że pierwszą funkcją matrycy jest rozcieńczanie i izolowanie cząsteczek analitu od siebie. Dzieje się to podczas odparowywania rozpuszczalnika i jednoczesnego tworzenia roztworu stałego. Następnie, po napromieniowaniu laserem, działa ona jako mediator dla absorpcji energii. Wybór odpowiedniej matrycy jest kluczem do sukcesu w MALDI. Generalnie, wysoko polarne anality lepiej współpracują z wysoko polarnymi matrycami, a niepolarne anality najlepiej łączyć z matrycami niepolarnymi. Jak pokazano w Tabeli 1, różne matryce były poszukiwane i szeroko stosowane. Obecnie, najczęściej stosowanymi matrycami są: kwas α-cyjano-4-hydroksycynamonowy, kwas 2,5-dihydroksybenzoesowy, kwas 3,5-dimetoksy-4-hydroksycynamonowy oraz 2,6-dihydroksyacetofenon.
Tabela 1. Matryce UV-MALDI (Gross J. H., 2006)
| Związek | Krótki skrót | Zastosowanie do |
|---|---|---|
| Kwas nikotynowy | NA | Peptydy, białka |
| Kwas pikolinowy | PA | Oligonukleotydy, DNA |
| 3-Hydroxypicolinic acid | HPA, 3-HPA | Oligonukleotydy, DNA |
| 3-Aminopikolinic acid | 3-APA | Oligonukleotydy, DNA |
| 6-Aza-2-thiothymine | ATT | Oligonukleotydy, DNA |
| Kwas 2,5-dihydroksybenzoesowy | DHB | Białka, oligosacharydy |
| Mieszaniny na bazie DHB | DHB/XY i super-DHB | Białka, oligosacharydy |
| 3-Aminochinolina | 3-AQ | Oligosacharydy |
| α-Kwas cyjano-4-hydroksycynamonowy | α-CHC, α-CHCA, 4-HCCA, CHCA | Peptydy, mniejsze białka, triacyloglicerole, liczne inne związki |
| 4-Chloro-α-cyano-cinnamic acid | ClCCA | Peptydy |
| 3,5-Dimetoksy-4-hydroksycynamonowy kwas | SA | Białka |
| 2-.(4-Hydroxyphenylazo) benzoic acid | HABA | Peptydy, białka, glikoproteiny, polistyren |
| 2-Merkaptobenzotiazol | MBT | Peptydy, białka, polimery syntetyczne |
| 5-Chloro-2-merkaptobenzotiazol | CMBT | Glikopeptydy, fosfopeptydy, i białka |
| 2,6-Dihydroxyacetophenone | DHAP | Glikopeptydy, fosfopeptydy, białka |
| 2,4,6-Trihydroksyacetofenon | THAP | Solidne oligonukleotydy |
| Dithranol (1,8,9-antracenetriol) | Nie | Polimery syntetyczne |
| 9-Nitroantracen | 9-NA | Fullereny i pochodne |
| Benzopiren | Nie | Fullereny i pochodne |
| 2-malonitryl | DCTB | Oligomery, polimery, dendrymery, małe cząsteczki |
Analizator czasu przelotu (TOF)
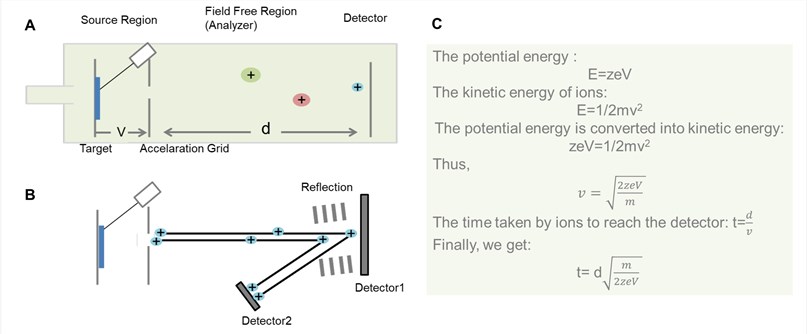
Figura 2. Ogólny schemat analizatora TOF. (A) Analizator TOF typu liner; (B) Analizator TOF typu reflector; (C) Proces wyznaczania czasu przejścia jonów przez obszar wolny od pola w analizatorze TOF typu liner.
- Zasada TOF
Jak pokazano na Rysunku 2, podstawowa zasada TOF polega na tym, że jony o różnych m/z są rozpraszane w czasie podczas ich lotu wzdłuż wolnej od pola ścieżki dryfu o znanej długości. Zakładając, że wszystkie jony rozpoczną swoją podróż w tym samym czasie lub przynajmniej w wystarczająco krótkim odstępie czasu, lżejsze z nich dotrą do detektora wcześniej niż cięższe.
- Linerowy analizator TOF i reflektronowy analizator TOF
Teoretycznie, wszystkie jony otrzymują taką samą początkową energię kinetyczną, tak że po dryfie wzdłuż obszaru wolnego od pola, jony o tym samym m/z dotrą do detektora w tym samym czasie. W praktyce jednak impuls nie jest odczuwany przez wszystkie jony z taką samą intensywnością i dlatego nie wszystkie jony o tych samych wartościach m/z osiągają idealne prędkości. Aby skorygować ten problem, na końcu strefy dryfu często stosuje się reflektron. Reflektron składa się z szeregu elektrod pierścieniowych o wysokim napięciu, które mogą odpychać jony z powrotem wzdłuż rurki prowadzącej, zwykle pod lekko przesuniętym kątem.
Iony o różnej energii kinetycznej penetrują reflektron na różne głębokości zanim zostaną wydalone z reflektronu w przeciwnym kierunku. Szybsze jony niosące większą energię kinetyczną pokonują dłuższą drogę niż wolniejsze, a zatem spędzają więcej czasu w reflektronie niż wolniejsze jony niosące mniejszą energię. W ten sposób do detektora trafiają jony o tej samej masie w (mniej więcej) tym samym czasie. Taka konstrukcja analizatora TOF znacznie zwiększyła jego rozdzielczość. Jednakże, reflektronowy analizator TOF nie jest odpowiedni dla analitów, które nie są wystarczająco stabilne, aby przetrwać pole elektryczne.
Proces spektrometrii mas MALDI-TOF
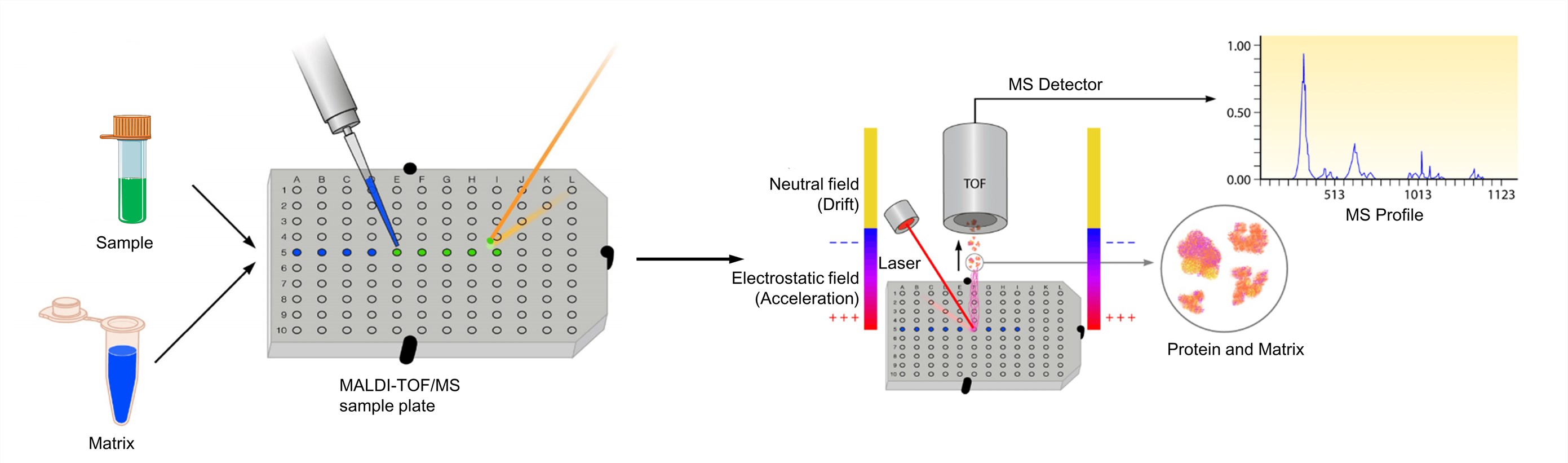
Rysunek 3. Proces spektrometrii mas MALDI-TOF (Clark A. E., et al.; 2013)
Analit powinien być rozpuszczalny do co najmniej około 0,1 mg/ml w jakimś rozpuszczalniku. Matrycę rozpuszcza się do uzyskania roztworu nasyconego lub o stężeniu około 10 mg/ml. Roztwór analitu jest następnie mieszany z roztworem matrycy. Dla zoptymalizowanych widm MALDI, stosunek molowy matrycy do analitu jest zwykle tak dobrany, aby mieścił się w zakresie od 1000: 1 do 100,000: 1. Następnie mieszanina jest nanoszona na metalową płytkę docelową w celu przeprowadzenia analizy. Po wysuszeniu, mieszanina próbki i matrycy współkrystalizuje i tworzy stały osad próbki osadzonej w matrycy. Płytka jest następnie ładowana do aparatu MALDI-TOF i analizowana przez oprogramowanie związane z danym systemem. MALDI prowadzi do sublimacji i jonizacji zarówno próbki jak i matrycy. Wygenerowane jony są rozdzielane w zależności od m/z przez analizator TOF, a reprezentacja spektralna tych jonów jest generowana i analizowana przez oprogramowanie MS, generując profil MS.
Zastosowanie spektrometrii mas MALDI-TOF
- Oznaczanie masy nienaruszonej
Oznaczanie masy nienaruszonej jest podstawowym i ważnym elementem charakteryzacji białek, ponieważ prawidłowa masa cząsteczkowa białka może wskazywać na jego nienaruszoną strukturę. MALDI, technika miękkiej jonizacji, jest odpowiednia dla białek, które mają tendencję do kruchości i fragmentacji podczas jonizacji innymi metodami jonizacji. Na wydajność MALDI-TOF MS mniejszy wpływ mają składniki buforu, detergenty i zanieczyszczenia. Ponadto, pozwala na określenie masy nienaruszonego białka z dokładnością wystarczającą do walidacji sekwencji (≤ 500 ppm). Po trawieniu białek, MALDI-TOF MS może być również wykorzystana do analizy otrzymanych peptydów w celu dalszego potwierdzenia sekwencji pierwotnej poprzez peptydowy odcisk mas (PMF).
- Peptide mass fingerprinting (PMF)
Spektrometria mas MALDI-TOF charakteryzuje się prostą obsługą, dobrą dokładnością mas, a także wysoką rozdzielczością i czułością. Z tego względu znajduje szerokie zastosowanie w proteomice do identyfikacji białek z prostych mieszanin metodą zwaną peptide mass fingerprinting, która jest często stosowana z dwuwymiarową elektroforezą żelową (2-DE). W tym podejściu, peptydy są generowane poprzez trawienie białek interesujących nas za pomocą enzymu specyficznego dla danej sekwencji, takiego jak trypsyna. Następnie peptydy są analizowane za pomocą spektrometrii mas MALDI-TOF w celu uzyskania mas peptydów. Masy doświadczalne są porównywane z bazą danych zawierającą teoretyczne masy peptydów z danego organizmu przy użyciu tej samej sekwencyjnie specyficznej proteazy.
- Analiza MALDI-TOF po rozpadzie źródła (PSD)
Spektrometry masowe MALDI-TOF wyposażone w reflektrony mogą analizować jony fragmentacyjne powstałe z jonów prekursorowych, które spontanicznie rozpadają się w locie. Takie jony są ogólnie określane jako jony metastabilne, a proces rozpadu w regionie wolnym od pola pomiędzy źródłem jonów a reflektronem jest powszechnie określany jako PSD. Jony fragmentaryczne PSD powstają w regionie wolnym od pola przed wejściem do reflektronu. Jony fragmentacyjne PSD mogą być rozdzielane, zbierane i rejestrowane na detektorze poprzez ciągłe zmiany napięcia reflektronu, tworząc widmo masowe PSD, które dostarcza bardzo bogatej i efektywnej informacji strukturalnej o strukturze pierwszorzędowej peptydów i białek. W badaniach proteomicznych, niektóre próbki białek rozdzielone metodą 2DE nie mogą być zidentyfikowane przez PMF lub wyniki identyfikacji nie są jednoznaczne. Funkcja sekwencjonowania PSD może być zastosowana do identyfikacji tych białek. Przy użyciu spektroskopii PSD, w połączeniu z przeszukiwaniem bazy danych, białka mogą być identyfikowane szybko i z wysoką specyficznością.
- Analiza oligonukleotydów
Wraz z rozwojem technik biologii molekularnej i technologii leków antysensownych kwasów nukleinowych, coraz więcej fragmentów oligonukleotydów zostało zsyntetyzowanych w celu wykorzystania ich jako startery, sondy i leki antysensowne. Konieczne jest szybkie wykrywanie tych fragmentów, aby określić, czy synteza jest zakończona i czy zsyntetyzowana sekwencja jest prawidłowa. Spektrometria mas, w tym MALDI-TOF-MS, jest do tego celu zdecydowanie najlepszym narzędziem. Analiza oligonukleotydów przy użyciu MALDI-TOF-MS była prosta, szybka, dokładna i czuła, co może być wykorzystane do określenia kompletnej sekwencji oligonukleotydu.
- Obrazowanie MALDI
Spektrometria MALDI-TOF może być wykorzystana do profilowania i obrazowania białek bezpośrednio z cienkich wycinków tkanek, znana jako MALDI-IMS (MALDI imaging mass spectrometry). Dostarcza ona specyficznych informacji o lokalnym składzie molekularnym, względnej obfitości i przestrzennym rozmieszczeniu peptydów i białek w analizowanym wycinku. MALDI-IMS może analizować wiele nieznanych związków w sekcjach tkanek biologicznych jednocześnie poprzez pojedynczy pomiar, który może uzyskać obrazowanie molekuł w tkance przy jednoczesnym zachowaniu integralności komórek i molekuł w tkankach.
Spektrometria mas MALDI-TOF może analizować szeroką gamę biomolekuł, takich jak peptydy, białka, węglowodany, oligonukleotydy, i tak dalej. Ze względu na fakt, że powstające jony mają niską energię wewnętrzną, wielką zaletą MALDI-TOF jest to, że proces miękkiej jonizacji umożliwia obserwację zjonizowanych cząsteczek przy niewielkiej fragmentacji analitów, co pozwala na identyfikację jonów molekularnych analitów, nawet w mieszaninach. Ponadto jest łatwa w użyciu i utrzymaniu, z szybką akwizycją danych. Wybór odpowiedniej substancji macierzowej jest ważny dla powodzenia spektrometrii mas MALDI-TOF.
W Creative Proteomics możemy świadczyć różne usługi w oparciu o nasze zaawansowane platformy spektrometrii mas MALDI-TOF, w tym:
- Określanie mas molekularnych
- Peptide Mass Fingerprinting (PMF)
- Imaging Mass Spectrometry (IMS)
1. Gross J H. Mass spectrometry: a textbook. Springer Science & Business Media, 2006.
2. Boesl U. Time – of – flight mass spectrometry: Wprowadzenie do podstaw. Mass spectrometry reviews, 2017, 36(1): 86-109.
3. Guerrera I C, Kleiner O. Zastosowanie spektrometrii mas w proteomice. Bioscience Reports, 2005, 25(1-2): 71-93.
4. Fuchs B, Schiller J. Application of MALD – TOF mass spectrometry in lipidomics. European Journal of Lipid Science and Technology, 2009, 111(1): 83-98.
5. Duncan M W, Roder H, Hunsucker S W. Quantitative matrix-assisted laser desorption/ionization mass spectrometry. Briefings in functional genomics and proteomics, 2008, 7(5): 355-370.
6. Kenny D J, Brown J M, Palmer M E, et al. A parallel approach to post source decay MALDI-TOF analysis. Journal of the American Society for Mass Spectrometry, 2006, 17(1): 60-66.