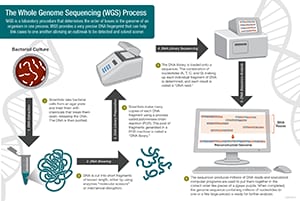
WGS Workflow: Larger View
Download pdf icon
Czym jest sekwencjonowanie całego genomu (WGS)?
Genom, czyli materiał genetyczny organizmu (bakterii, wirusa, ziemniaka, człowieka) składa się z DNA. Każdy organizm ma unikalną sekwencję DNA, która składa się z zasad (A, T, C i G). Jeśli znasz sekwencję zasad w danym organizmie, zidentyfikowałeś jego unikalny odcisk palca DNA, czyli wzór. Określenie kolejności zasad nazywa się sekwencjonowaniem. Sekwencjonowanie całych genomów jest procedurą laboratoryjną, która pozwala określić kolejność zasad w genomie organizmu w jednym procesie.
Jak działa sekwencjonowanie całych genomów?
Naukowcy przeprowadzają sekwencjonowanie całych genomów, wykonując następujące cztery główne kroki:
- Ścinanie DNA: Naukowcy zaczynają od użycia molekularnych nożyczek do cięcia DNA, które składa się z milionów zasad: A, C, T i G, na kawałki, które są wystarczająco małe, aby maszyna do sekwencjonowania mogła je odczytać.
- Kodowanie paskowe DNA: Naukowcy dodają małe kawałki znaczników DNA, lub kody kreskowe, aby zidentyfikować, który kawałek ściętego DNA należy do której bakterii. Jest to podobne do tego, jak kod kreskowy identyfikuje produkt w sklepie spożywczym.
- Sekwencjonowanie całego genomu: Zakodowane paskowo DNA z wielu bakterii jest łączone i umieszczane w sekwenatorze całego genomu. Sekwenator identyfikuje litery A, C, T i G, lub zasady, które tworzą każdą sekwencję bakterii. Sekwenator używa kodu kreskowego do śledzenia, które bazy należą do której bakterii.
- Analiza danych: Naukowcy używają narzędzi do analizy komputerowej, aby porównać sekwencje bakterii i zidentyfikować różnice. Liczba różnic może powiedzieć naukowcom, jak blisko spokrewnione są bakterie i jak prawdopodobne jest, że są częścią tej samej epidemii.

Jak sekwencjonowanie całego genomu zmieni wykrywanie chorób?
Sekwencjonowanie całego genomu zapewnia bardziej szczegółowe i precyzyjne dane do identyfikacji ognisk choroby niż obecna standardowa technika, którą stosuje PulseNet, elektroforeza w żelu o pulsacyjnym polu magnetycznym (PFGE). Zamiast mieć możliwość porównywania genomów bakterii tylko na podstawie 15-30 pasm, które pojawiają się we wzorze PFGE, mamy teraz miliony baz do porównania. To tak, jakbyśmy porównywali wszystkie słowa w książce (WGS), zamiast tylko liczbę rozdziałów (PFGE), aby sprawdzić, czy książki są takie same czy różne. Używając sekwencjonowania całego genomu, odkryliśmy, że niektóre bakterie, które wydawały się być różne przy użyciu PFGE, w rzeczywistości pochodzą z tego samego źródła. Pomogło to w szybszym rozwiązaniu niektórych ognisk epidemicznych.
Sekwencjonowanie całego genomu jest szybkim i niedrogim sposobem uzyskania wysokopoziomowych informacji o bakteriach przy użyciu tylko jednego testu. Obecnie proces pełnego scharakteryzowania bakterii wymaga przeprowadzenia przez dwóch lub więcej naukowców czterech lub więcej oddzielnych testów, w tym PFGE. WGS znacznie poprawi efektywność sposobu, w jaki PulseNet prowadzi nadzór.
PulseNet aktywnie waliduje technologię sekwencjonowania następnej generacji (NGS), jak również opracowuje, ocenia i wdraża narzędzia potrzebne do analizy danych.
Podejmowanie wyzwań związanych z sekwencjonowaniem całego genomu.
W 2013 roku CDC zaczęło używać sekwencjonowania całego genomu do wykrywania ognisk wywołanych przez śmiertelną bakterię Listeria. Od tego czasu metoda ta pozwoliła naukowcom na:
- Wykrywanie większej liczby skupisk zachorowań wywołanych przez bakterie Listeria
- Rozwiązanie większej liczby ognisk Listeria, gdy są one jeszcze małe
- Połączenie chorych pacjentów z prawdopodobnymi źródłami żywności
- Zidentyfikowanie nowych źródeł żywności Listeria, takich jak jabłka karmelowe i lody
Dowiedz się więcej o tym, jak projekt sekwencjonowania całego genomu Listeria usprawnił wykrywanie i badanie ognisk chorób przenoszonych drogą pokarmową.
CDC szybko rozszerza zastosowanie sekwencjonowania całych genomów w laboratoriach stanowych, a naukowcy wkrótce zaczną wykorzystywać sekwencjonowanie całych genomów do badania ognisk innych patogenów przenoszonych przez żywność, takich jak Campylobacter, E. coli produkująca toksynę Shiga (STEC) i Salmonella. Inicjatywa Advanced Molecular Detection (AMD) firmy CDC częściowo sfinansowała ekspansję WGS w czasie rzeczywistym dla bezpieczeństwa żywności.
Dzięki współpracy z inicjatywą AMD firmy CDC oraz programem bezpieczeństwa żywności, PulseNet tworzy strukturę wspierającą przejście na sekwencjonowanie całego genomu, w tym szkolenie mikrobiologów zdrowia publicznego do wykonywania sekwencjonowania, zakup materiałów do sekwencjonowania oraz aktualizację systemów do analizy danych. Działania te są kluczowe dla uruchomienia sekwencjonowania całych genomów w laboratoriach zdrowia publicznego i poprawy nadzoru nad ogniskami chorób przenoszonych drogą pokarmową oraz trendami w zakażeniach przenoszonych drogą pokarmową i oporności na antybiotyki.
W miarę rozszerzania się zastosowania sekwencjonowania całych genomów krajowe systemy nadzoru i infrastruktura laboratoryjna CDC muszą dotrzymywać kroku zmieniającej się technologii. Dzięki modernizacji, CDC i jego partnerzy w dziedzinie zdrowia publicznego mogą nadal skutecznie wykrywać, reagować i powstrzymywać choroby zakaźne. Razem możemy zapewnić szybkie i mniej kosztowne diagnozy dla osób indywidualnych oraz dowody potrzebne do szybkiego rozwiązywania i zapobiegania epidemiom chorób przenoszonych przez żywność.