Flux de travail WGS : Vue plus grande
Télécharger l’icône pdf
Qu’est-ce que le séquençage du génome entier (WGS)?
Le génome, ou matériel génétique, d’un organisme (bactérie, virus, pomme de terre, humain) est constitué d’ADN. Chaque organisme possède une séquence d’ADN unique qui est composée de bases (A, T, C et G). Si vous connaissez la séquence des bases d’un organisme, vous avez identifié son empreinte génétique unique, ou modèle. La détermination de l’ordre des bases est appelée séquençage. Le séquençage du génome entier est une procédure de laboratoire qui détermine l’ordre des bases dans le génome d’un organisme en un seul processus.
Comment fonctionne le séquençage du génome entier ?
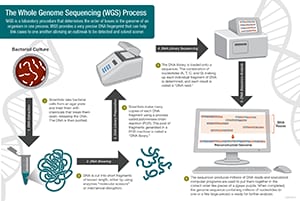
Les scientifiques réalisent le séquençage du génome entier en suivant ces quatre étapes principales :
- Cisaillement de l’ADN : Les scientifiques commencent par utiliser des ciseaux moléculaires pour couper l’ADN, qui est composé de millions de bases : A, C, T et G, en morceaux suffisamment petits pour que la machine de séquençage puisse les lire.
- Codage à barres de l’ADN : Les scientifiques ajoutent de petits morceaux d’étiquettes d’ADN, ou codes-barres, pour identifier quel morceau d’ADN cisaillé appartient à quelle bactérie. Cela ressemble à la façon dont un code-barres identifie un produit dans une épicerie.
- Séquençage du génome entier : L’ADN à code-barres de plusieurs bactéries est combiné et placé dans le séquenceur du génome entier. Le séquenceur identifie les A, C, T et G, ou bases, qui composent chaque séquence bactérienne. Le séquenceur utilise le code-barres pour garder la trace de quelles bases appartiennent à quelles bactéries.
- Analyse des données : Les scientifiques utilisent des outils d’analyse informatique pour comparer les séquences bactériennes et identifier les différences. Le nombre de différences peut indiquer aux scientifiques à quel point les bactéries sont étroitement liées et quelle est la probabilité qu’elles fassent partie de la même épidémie.

Comment le séquençage du génome entier va-t-il transformer la détection des maladies ?
Le séquençage du génome entier fournit des données plus détaillées et plus précises pour identifier les épidémies que la technique standard actuelle utilisée par PulseNet, l’électrophorèse en champ pulsé (PFGE). Au lieu de ne pouvoir comparer les génomes bactériens qu’à partir des 15 à 30 bandes qui apparaissent dans un schéma PFGE, nous avons maintenant des millions de bases à comparer. C’est comme si l’on comparait tous les mots d’un livre (WGS), au lieu de se limiter au nombre de chapitres (PFGE), pour voir si les livres sont identiques ou différents. Grâce au séquençage du génome entier, nous avons découvert que certaines bactéries qui semblaient différentes en utilisant la PFGE provenaient en fait de la même source. Cela a permis de résoudre plus rapidement certaines épidémies.
Le séquençage du génome entier est un moyen rapide et abordable d’obtenir des informations de haut niveau sur les bactéries en utilisant un seul test. Actuellement, le processus de caractérisation complète des bactéries nécessite deux scientifiques ou plus pour effectuer quatre tests distincts ou plus, y compris la PFGE. Le WGS améliorera considérablement l’efficacité de la façon dont PulseNet mène la surveillance.
PulseNet valide activement la technologie de séquençage de nouvelle génération (NGS) ainsi que le développement, l’évaluation et la mise en œuvre des outils nécessaires pour analyser les données.
Répondre aux défis du séquençage du génome entier.
En 2013, les CDC ont commencé à utiliser le séquençage du génome entier pour détecter les épidémies causées par la bactérie mortelle Listeria. Depuis lors, cette méthode a permis aux scientifiques de :
- Détecter davantage de grappes de maladies causées par Listeria
- Résoudre davantage d’épidémies de Listeria alors qu’elles sont encore petites
- Lier les patients malades à des sources alimentaires probables
- Identifier de nouvelles sources alimentaires de Listeria, comme les pommes au caramel et la crème glacée
En savoir plus sur la façon dont le projet de séquençage du génome entier de Listeria a amélioré la détection et l’investigation des épidémies d’origine alimentaire.
Le CDC étend rapidement l’utilisation du séquençage du génome entier dans les laboratoires d’État, et les scientifiques commenceront bientôt à utiliser le séquençage du génome entier pour les enquêtes sur les épidémies d’autres agents pathogènes d’origine alimentaire, tels que Campylobacter, E. coli producteur de toxines de Shiga (STEC) et Salmonella. L’initiative de détection moléculaire avancée (AMD) des CDC a partiellement financé l’expansion du WGS en temps réel pour la sécurité alimentaire.
Grâce à la collaboration avec l’initiative AMD des CDC et le programme de sécurité alimentaire, PulseNet met en place la structure nécessaire pour soutenir un passage au séquençage du génome entier, y compris la formation des microbiologistes de la santé publique pour effectuer le séquençage, l’achat de fournitures de séquençage et la mise à jour des systèmes d’analyse des données. Ces activités sont essentielles au lancement du séquençage du génome entier dans les laboratoires de santé publique et à l’amélioration de la surveillance des épidémies de maladies d’origine alimentaire et des tendances en matière d’infections d’origine alimentaire et de résistance aux antibiotiques.
Au fur et à mesure que l’utilisation du séquençage du génome entier se développe, les systèmes de surveillance nationaux et l’infrastructure des laboratoires du CDC doivent suivre l’évolution de la technologie. Avec la modernisation, le CDC et ses partenaires de la santé publique peuvent continuer à détecter, à répondre et à arrêter avec succès les maladies infectieuses. Ensemble, nous pouvons assurer des diagnostics rapides et moins coûteux pour les individus et les preuves nécessaires pour résoudre et prévenir rapidement les épidémies d’origine alimentaire.
Les systèmes de surveillance et les infrastructures de laboratoire du CDC doivent être modernisés.