La spectrométrie de masse est une technique d’analyse dans laquelle les échantillons sont ionisés en molécules chargées et le rapport de leur masse à charge (m/z) peut être mesuré. Dans la spectrométrie de masse MALDI-TOF, la source d’ions est la désorption/ionisation laser assistée par matrice (MALDI), et l’analyseur de masse est l’analyseur à temps de vol (TOF).
MALDI
La MALDI est une ionisation douce qui implique un laser frappant une matrice de petites molécules pour faire passer les molécules d’analyte dans la phase gazeuse sans les fragmenter ou les décomposer. Certaines biomolécules sont trop grosses et peuvent se décomposer lorsqu’elles sont chauffées, et les techniques traditionnelles fragmentent ou détruisent les macromolécules. Le MALDI est approprié pour analyser des biomolécules comme les peptides, les lipides, les saccharides ou d’autres macromolécules organiques.
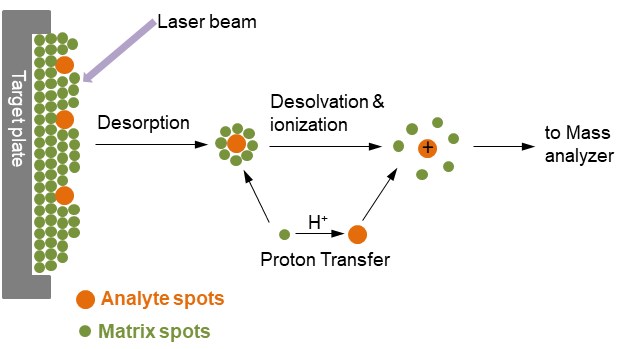
Figure 1. Ionisation d’analytes par MALDI
- Le principe du MALDI
Dans la figure 1, l’analyte est noyé dans un très grand excès d’un composé matriciel déposé sur une surface solide appelée cible, généralement constituée d’un métal conducteur et comportant des spots pour plusieurs échantillons différents à appliquer. Après une très brève impulsion laser, le point irradié est rapidement chauffé et devient vibratoirement excité. Les molécules de la matrice ablatées énergétiquement de la surface de l’échantillon, absorbent l’énergie laser et entraînent les molécules de l’analyte dans la phase gazeuse également. Pendant le processus d’ablation, les molécules d’analyte sont généralement ionisées en étant protonées ou déprotonées avec les molécules de matrice proches. Le format d’ionisation MALDI le plus courant est que les molécules d’analyte portent une seule charge positive.
- Types de laser couramment utilisés en MALDI
Des lasers de longueurs d’onde ultraviolettes (UV) et infrarouges (IR) sont utilisés, mais les lasers UV sont de loin les sources de lumière les plus importantes en MALDI analytique. Parmi eux, les lasers à azote et les lasers Nd : Yag triplés ou quadruplés en fréquence sont souvent utilisés pour la majorité des applications. L’IR-MALDI est dominé par les lasers Er:Yag tandis que les lasers TEA-CO2 sont rarement utilisés.
- Substance de matrice MALDI couramment utilisée
On pense que la première fonction de la matrice consiste essentiellement à diluer et à isoler les molécules d’analyte les unes des autres. Cela se produit pendant l’évaporation du solvant et la formation concomitante d’une solution solide. Ensuite, lors de l’irradiation laser, elle fonctionne comme un médiateur pour l’absorption d’énergie. Le choix de la bonne matrice est la clé du succès en MALDI. En général, les analytes hautement polaires fonctionnent mieux avec des matrices hautement polaires, et les analytes non polaires sont de préférence combinés avec des matrices non polaires. Comme le montre le tableau 1, différentes matrices ont été recherchées et largement utilisées. Actuellement, les matrices les plus utilisées sont l’acide α-cyano-4-hydroxycinnamique, l’acide 2,5-dihydroxybenzoïque, l’acide 3,5-diméthoxy-4-hydroxycinnamique et la 2,6-dihydroxyacétophénone.
Tableau 1. Matrices UV-MALDI (Gross J. H., 2006)
| Composé | Acronyme | Application à |
|---|---|---|
| Acide nicotinique | NA | Peptides, protéines |
| Acide picolinique | PA | Oligonucléotides, ADN |
| Acide 3-hydroxypicolinique | HPA, 3-HPA | Oligonucléotides, ADN |
| Acide 3-aminopicolinique | 3-APA | Oligonucléotides, ADN |
| 6-Aza-2-thiothymine | ATT | Oligonucléotides, ADN |
| Acide 2,5-dihydroxybenzoïque | DHB | Protéines, oligosaccharides |
| Mélanges à base de DHB | DHB/XY et super-DHB | Protéines, oligosaccharides |
| 3-Aminoquinoléine | 3-AQ | Oligosaccharides |
| Acideα-Cyano-4-hydroxycinnamique | α-CHC, α-CHCA, 4-HCCA, CHCA | Peptides, protéines plus petites, triacylglycérols, nombreux autres composés |
| 4-Chloro-α-cyano-acide cinnamique | ClCCA | Peptides |
| 3,5-Dimethoxy-4-hydroxycinnamic acid | SA | Proteines | 2-(4-Hydroxyphenylazo) benzoic acid | HABA | Peptides, protéines, glycoprotéines, polystyrène |
| 2-Mercaptobenzothiazole | MBT | Peptides, protéines, polymères synthétiques |
| 5-Chloro-2-mercaptobenzothiazole | CMBT | Glycopeptides, phosphopeptides, et protéines |
| 2,6-Dihydroxyacétophénone | DHAP | Glycopeptides, phosphopeptides, protéines |
| 2,4,6-Trihydroxyacétophénone | THAP | Oligonucléotides sur support solide | Dithranol (1,8,9-anthracénétriol) | None | Polymères synthétiques |
| 9-Nitroanthracène | 9-NA | Fullerènes et dérivés |
| Benzopyrène | None | Fullerènes et dérivés |
| 2-malonitrile | DCTB | Oligomères, polymères, dendrimères, petites molécules |
Analyseur à temps de vol (TOF)
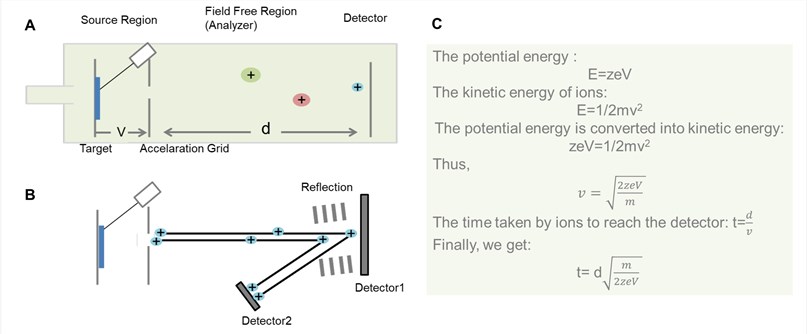
Figure 2. Schéma général d’un analyseur TOF. (A) Analyseur TOF à doublure ; (B) Analyseur TOF à réflecteur ; (C) Le processus de dérivation du temps de passage des ions dans la région sans champ dans l’analyseur TOF à doublure.
- Le principe de la TOF
Comme le montre la figure 2, le principe de base de la TOF est que les ions de différents m/z sont dispersés dans le temps pendant leur vol le long d’un chemin de dérive sans champ de longueur connue. À condition que tous les ions commencent leur voyage en même temps ou au moins dans un intervalle de temps suffisamment court, les plus légers arriveront plus tôt au détecteur que les plus lourds.
- Analyseur TOF à liner et analyseur TOF à réflectron
Théoriquement, tous les ions reçoivent la même énergie cinétique initiale, de sorte qu’après avoir dérivé le long de la région sans champ, les ions de même m/z au détecteur au moment. Cependant, dans la pratique, l’impulsion n’est pas ressentie par tous les ions avec la même intensité et donc tous les ions de même valeur m/z n’atteignent pas leur vitesse idéale. Pour corriger ce problème, on applique souvent un réflecteur à l’extrémité de la zone de dérive. Le réflectron est constitué d’une série d’électrodes annulaires à haute tension, qui peuvent repousser les ions le long du tube de vol généralement à un angle légèrement déplacé.
Les ions d’énergie cinétique différente pénètrent dans le réflectron à des profondeurs différentes avant d’être expulsés du réflectron dans la direction opposée. Les ions plus rapides transportant plus d’énergie cinétique parcourent un chemin plus long que les ions plus lents, et passent donc plus de temps dans le réflectron que les ions plus lents transportant moins d’énergie. De cette manière, le détecteur reçoit des ions de même masse au même moment (ou presque). Ainsi, cette conception de l’analyseur de masse TOF a permis d’augmenter considérablement sa résolution. Cependant, l’analyseur TOF à réflecteur ne convient pas aux analytes qui ne sont pas assez stables pour survivre au champ électrique.
Le processus de spectrométrie de masse MALDI-TOF
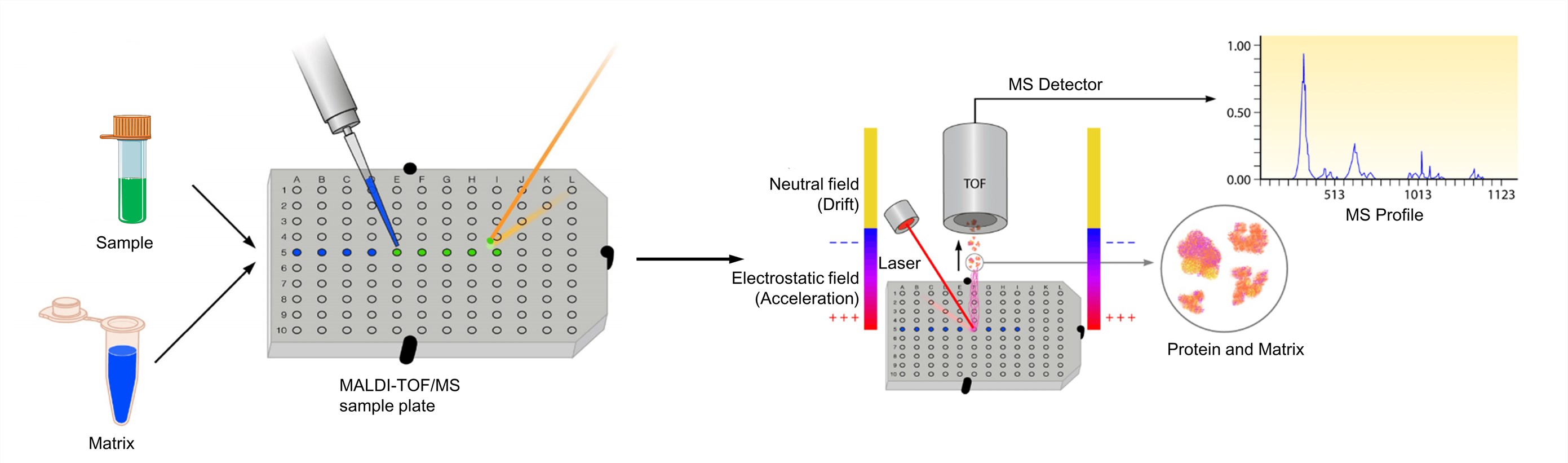
Figure 3. Le processus de spectrométrie de masse MALDI-TOF (Clark A. E., et al. ; 2013)
L’analyte doit être soluble à au moins environ 0,1 mg/ml dans un certain solvant. Et la matrice est dissoute pour donner soit une solution saturée, soit une concentration d’environ 10 mg/ml. La solution de l’analyte est ensuite mélangée à celle de la matrice. Pour optimiser les spectres MALDI, le rapport molaire matrice/analyte est normalement ajusté de manière à se situer dans la plage de 1000 : 1 à 100 000 : 1. Le mélange est ensuite déposé sur une plaque cible métallique pour l’analyse. Après séchage, le mélange de l’échantillon et de la matrice se co-cristallise et forme un dépôt solide d’échantillon noyé dans la matrice. La plaque est ensuite chargée dans l’instrument MALDI-TOF et analysée par le logiciel associé au système respectif. Le MALDI entraîne la sublimation et l’ionisation de l’échantillon et de la matrice. Ces ions générés sont séparés en fonction du m/z par un analyseur TOF, et une représentation spectrale de ces ions est générée et analysée par le logiciel MS, générant un profil MS.
Application de la spectrométrie de masse MALDI-TOF
- Détermination de la masse intacte
La détermination de la masse intacte est fondamentale et importante pour la caractérisation des protéines, car la masse moléculaire correcte d’une protéine peut indiquer la structure intacte. Le MALDI, une technique d’ionisation douce, est adapté aux protéines qui ont tendance à être fragiles et à se fragmenter lorsqu’elles sont ionisées par d’autres méthodes d’ionisation. Les performances du MALDI-TOF MS sont moins affectées par les composants du tampon, les détergents et les contaminants. En outre, elle permet de déterminer la masse des protéines intactes avec une précision suffisante (≤ 500 ppm) pour la validation des séquences. Après la digestion des protéines, la spectrométrie de masse MALDI-TOF peut également être utilisée pour analyser les peptides obtenus en vue d’une confirmation supplémentaire de la séquence primaire par empreinte de masse peptidique.
- Imprimé de masse peptidique (PMF)
La spectrométrie de masse MALDI-TOF présente une opération simple, une bonne précision de masse, ainsi qu’une résolution et une sensibilité élevées. Par conséquent, elle est largement utilisée en protéomique pour identifier les protéines à partir de mélanges simples par une méthode appelée empreinte de masse peptidique, qui sont souvent utilisées avec l’électrophorèse sur gel bidimensionnelle (2-DE). Dans cette approche, les peptides sont générés en digérant les protéines d’intérêt avec une enzyme spécifique à la séquence, comme la trypsine. Puis les peptides sont analysés par spectrométrie de masse MALDI-TOF pour obtenir les masses des peptides. Les masses expérimentales sont comparées à une base de données contenant les masses peptidiques théoriques d’un organisme donné avec la même protéase spécifique de la séquence.
- Analyse MALDI-TOF par décroissance post-source (PSD)
Les spectromètres de masse MALDI-TOF équipés de réflecteurs peuvent analyser les ions fragments produits à partir d’ions précurseurs qui se décomposent spontanément dans le vol. De tels ions sont généralement appelés ions métastables, et le processus de décomposition dans la région sans champ entre la source d’ions et le réflectron est communément appelé PSD. Les ions fragments PSD sont formés dans la région sans champ avant d’entrer dans le réflecteur. Les ions fragments PSD peuvent être séparés, collectés et enregistrés sur le détecteur en changeant continuellement la tension du réflecteur pour former un spectre de masse PSD qui fournit des informations structurelles très riches et efficaces pour la structure primaire des peptides et des protéines. Dans l’étude de la protéomique, certains échantillons de protéines séparés par 2DE ne peuvent pas être identifiés par PSD ou les résultats de l’identification ne sont pas clairs. La fonction de séquençage PSD peut être appliquée à l’identification de ces protéines. En utilisant la spectroscopie PSD, combinée à une recherche dans la base de données, les protéines peuvent être identifiées rapidement et avec une grande spécificité.
- Analyse des oligonucléotides
Avec le développement des techniques de biologie moléculaire et des technologies de médicaments à base d’acide nucléique antisens, de plus en plus de fragments d’oligonucléotides ont été synthétisés pour être utilisés comme amorces, sondes et médicaments antisens. Il est tout à fait nécessaire de détecter rapidement ces fragments pour déterminer si la synthèse est terminée et si la séquence synthétisée est correcte. La spectrométrie de masse, y compris le MALDI-TOF-MS, est de loin le meilleur moyen d’y parvenir. L’analyse des oligonucléotides à l’aide de la MALDI-TOF-MS était simple, rapide, précise et sensible, ce qui peut être utilisé pour déterminer la séquence complète des oligonucléotides.
- Imagerie MALDI
La MALDI-TOF peut être utilisée dans le profilage et l’imagerie des protéines directement à partir de fines sections de tissus, connue sous le nom de spectrométrie de masse d’imagerie MALDI (MALDI-IMS). Elle fournit des informations spécifiques sur la composition moléculaire locale, l’abondance relative et la distribution spatiale des peptides et des protéines dans la section analysée. La MALDI-IMS peut analyser plusieurs composés inconnus dans des sections de tissus biologiques simultanément par une seule mesure qui peut obtenir une imagerie moléculaire du tissu tout en maintenant l’intégrité des cellules et des molécules dans les tissus.
La spectrométrie de masse MALDI-TOF peut analyser une grande variété de biomolécules, telles que les peptides, les protéines, les glucides, les oligonucléotides, etc. Étant donné que les ions formés ont une faible énergie interne, un grand avantage du MALDI-TOF est que le processus d’ionisation douce permet d’observer les molécules ionisées avec peu de fragmentation des analytes, ce qui permet d’identifier les ions moléculaires des analytes, même dans les mélanges. De plus, il est facile à utiliser et à entretenir, avec une acquisition rapide des données. Le choix de la substance matricielle appropriée est important pour une spectrométrie de masse MALDI-TOF réussie.
À Creative Proteomics, nous pouvons fournir divers services basés sur nos plateformes avancées de spectrométrie de masse MALDI-TOF, notamment :
- Détermination de la masse moléculaire
- Peptide Mass Fingerprinting (PMF)
- Spectrométrie de masse par imagerie (IMS)
1. Gross J H. Spectrométrie de masse : un manuel. Springer Science & Business Media, 2006.
2. Boesl U. Time – of – flight mass spectrometry : Introduction aux principes de base. Mass spectrometry reviews, 2017, 36(1) : 86-109.
3. Guerrera I C, Kleiner O. Application de la spectrométrie de masse en protéomique. Bioscience Reports, 2005, 25(1-2) : 71-93.
4. Fuchs B, Schiller J. Application de la spectrométrie de masse MALD – TOF en lipidomique. European Journal of Lipid Science and Technology, 2009, 111(1) : 83-98.
5. Duncan M W, Roder H, Hunsucker S W. Quantitative matrix-assisted laser desorption/ionization mass spectrometry. Briefings in functional genomics and proteomics, 2008, 7(5) : 355-370.
6. Kenny D J, Brown J M, Palmer M E, et al. A parallel approach to post source decay MALDI-TOF analysis. Journal of the American Society for Mass Spectrometry, 2006, 17(1) : 60-66.