WGS Workflow: Visualizzazione ingrandita
Scaricare l’icona pdf
Che cos’è il sequenziamento del genoma intero (WGS)?
Il genoma, o materiale genetico, di un organismo (batteri, virus, patata, uomo) è costituito da DNA. Ogni organismo ha una sequenza unica di DNA che è composta da basi (A, T, C e G). Se conosci la sequenza delle basi in un organismo, hai identificato la sua impronta digitale unica del DNA, o modello. Determinare l’ordine delle basi è chiamato sequenziamento. Il sequenziamento del genoma intero è una procedura di laboratorio che determina l’ordine delle basi nel genoma di un organismo in un unico processo.
Come funziona il sequenziamento del genoma intero?
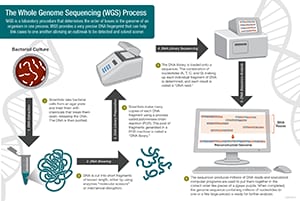
Gli scienziati conducono il sequenziamento del genoma intero seguendo questi quattro passi principali:
- Taglio del DNA: Gli scienziati iniziano usando delle forbici molecolari per tagliare il DNA, che è composto da milioni di basi: A, C, T e G, in pezzi abbastanza piccoli da essere letti dalla macchina di sequenziamento.
- DNA bar-coding: Gli scienziati aggiungono piccoli pezzi di DNA, o codici a barre, per identificare quale pezzo di DNA tosato appartiene a quale batterio. Questo è simile a come un codice a barre identifica un prodotto in un negozio di alimentari.
- Sequenziamento del genoma intero: Il DNA con codice a barre da più batteri viene combinato e messo nel sequenziatore del genoma intero. Il sequenziatore identifica le A, C, T e G, o basi, che compongono ogni sequenza batterica. Il sequenziatore usa il codice a barre per tenere traccia di quali basi appartengono a quale batterio.
- Analisi dei dati: Gli scienziati usano strumenti di analisi al computer per confrontare le sequenze batteriche e identificare le differenze. Il numero di differenze può dire agli scienziati quanto strettamente correlati siano i batteri e quanto sia probabile che facciano parte dello stesso focolaio.

In che modo il sequenziamento del genoma intero trasformerà la rilevazione delle malattie?
Il sequenziamento del genoma intero fornisce dati più dettagliati e precisi per identificare i focolai rispetto all’attuale tecnica standard utilizzata da PulseNet, l’elettroforesi in gel a campo pulsato (PFGE). Invece di avere solo la capacità di confrontare i genomi batterici utilizzando 15-30 bande che appaiono in un modello PFGE, ora abbiamo milioni di basi da confrontare. È come confrontare tutte le parole di un libro (WGS), invece che solo il numero di capitoli (PFGE), per vedere se i libri sono uguali o diversi. Usando il sequenziamento del genoma intero, abbiamo scoperto che alcuni batteri che sembravano diversi usando la PFGE sono in realtà della stessa fonte. Questo ha aiutato a risolvere alcuni focolai prima.
Il sequenziamento del genoma intero è un modo veloce e conveniente per ottenere informazioni di alto livello sui batteri usando solo un test. Attualmente, il processo per caratterizzare completamente i batteri richiede due o più scienziati per eseguire quattro o più test separati tra cui PFGE. WGS migliorerà notevolmente l’efficienza di come PulseNet conduce la sorveglianza.
PulseNet sta attivamente convalidando la tecnologia di sequenziamento di prossima generazione (NGS) così come lo sviluppo, la valutazione e l’implementazione degli strumenti necessari per analizzare i dati.
Rispondere alle sfide del sequenziamento del genoma intero.
Nel 2013, CDC ha iniziato a utilizzare sequenziamento del genoma intero per rilevare focolai causati dal batterio mortale Listeria. Da allora, questo metodo ha permesso agli scienziati di:
- Rilevare più gruppi di malattie da Listeria
- Risolvere più focolai di Listeria quando sono ancora piccoli
- Collegare i pazienti malati alle probabili fonti alimentari
- Identificare nuove fonti alimentari di Listeria, come le mele caramellate e il gelato
Scopri di più su come il Listeria Whole Genome Sequencing Project ha migliorato l’individuazione e l’indagine dei focolai di origine alimentare.
Il CDC sta rapidamente espandendo l’uso del sequenziamento del genoma intero nei laboratori statali, e gli scienziati inizieranno presto ad usare il sequenziamento del genoma intero per le indagini sui focolai di altri patogeni di origine alimentare, come Campylobacter, E. coli produttore della tossina Shiga (STEC) e Salmonella. L’iniziativa Advanced Molecular Detection (AMD) del CDC ha parzialmente finanziato l’espansione del WGS in tempo reale per la sicurezza alimentare.
Attraverso la collaborazione con l’iniziativa AMD del CDC e il programma di sicurezza alimentare, PulseNet sta stabilendo la struttura per sostenere un passaggio al sequenziamento del genoma intero, compresa la formazione di microbiologi della sanità pubblica per eseguire il sequenziamento, l’acquisto di forniture di sequenziamento e l’aggiornamento dei sistemi per l’analisi dei dati. Queste attività sono fondamentali per lanciare il sequenziamento del genoma intero nei laboratori di salute pubblica e migliorare la sorveglianza per i focolai di malattie di origine alimentare e le tendenze nelle infezioni di origine alimentare e la resistenza agli antibiotici.
Come l’uso del sequenziamento del genoma intero si espande, i sistemi di sorveglianza nazionale del CDC e le infrastrutture di laboratorio devono tenere il passo con la tecnologia che cambia. Con la modernizzazione, CDC e i suoi partner di salute pubblica possono continuare a rilevare, rispondere e fermare con successo le malattie infettive. Insieme, possiamo garantire diagnosi rapide e meno costose per gli individui e le prove necessarie per risolvere e prevenire rapidamente le epidemie di origine alimentare.