La spettrometria di massa è una tecnica analitica in cui i campioni sono ionizzati in molecole cariche e il rapporto della loro massa/carica (m/z) può essere misurato. Nella spettrometria di massa MALDI-TOF, la sorgente ionica è la desorbimento/ionizzazione laser assistita da matrice (MALDI) e l’analizzatore di massa è l’analizzatore TOF (time-of-flight).
MALDI
MALDI è una ionizzazione morbida che comporta un laser che colpisce una matrice di piccole molecole per far entrare le molecole dell’analita nella fase gassosa senza frammentarle o decomporle. Alcune biomolecole sono troppo grandi e possono decomporsi se riscaldate, e le tecniche tradizionali frammentano o distruggono le macromolecole. MALDI è appropriato per analizzare biomolecole come peptidi, lipidi, saccaridi, o altre macromolecole organiche.
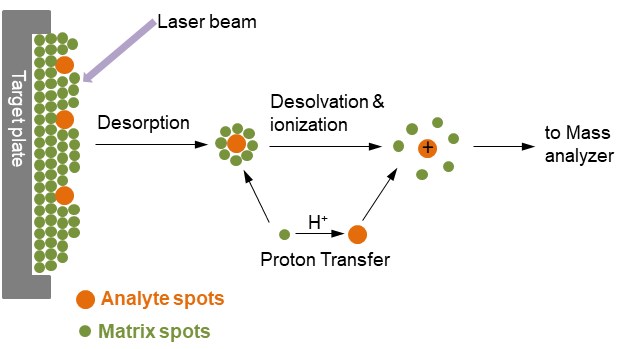
Figura 1. Ionizzazione di analiti tramite MALDI
- Il principio del MALDI
Nella figura 1, l’analita è incorporato in un eccesso molto grande di un composto matrice depositato su una superficie solida chiamata bersaglio, di solito fatta di un metallo conduttore e con punti per diversi campioni da applicare. Dopo un impulso laser molto breve, lo spot irradiato viene rapidamente riscaldato e viene eccitato vibrazionalmente. Le molecole della matrice, energicamente ablate dalla superficie del campione, assorbono l’energia del laser e portano le molecole dell’analita anche nella fase gassosa. Durante il processo di ablazione, le molecole dell’analita sono solitamente ionizzate essendo protonate o deprotonate con le molecole della matrice vicine. Il formato di ionizzazione MALDI più comune è che le molecole di analita portino una singola carica positiva.
- Tipi di laser comunemente usati in MALDI
Sono in uso laser sia di lunghezza d’onda ultravioletta (UV) che infrarossa (IR), ma i laser UV sono di gran lunga le fonti di luce più importanti nel MALDI analitico. Tra questi, i laser ad azoto e i laser Nd: Yag triplicati o quadruplicati in frequenza servono spesso per la maggior parte delle applicazioni. L’IR-MALDI è dominato dai laser Er:Yag mentre i laser TEA-CO2 sono usati raramente.
- Sostanza matrice MALDI comunemente usata
Si ritiene che la prima funzione della matrice sia essenzialmente quella di diluire e isolare le molecole di analita le une dalle altre. Questo avviene durante l’evaporazione del solvente e la concomitante formazione di una soluzione solida. Poi, all’irradiazione laser, funziona come mediatore per l’assorbimento dell’energia. La scelta della matrice giusta è la chiave del successo nella MALDI. In generale, gli analiti altamente polari lavorano meglio con matrici altamente polari, e gli analiti non polari sono preferibilmente combinati con matrici non polari. Come mostrato nella tabella 1, diverse matrici sono state ricercate e ampiamente utilizzate. Attualmente, le matrici più comunemente usate sono l’acido α-ciano-4-idrossicinnamico, l’acido 2,5-diidrossibenzoico, l’acido 3,5-dimetossi-4-idrossicinnamico e il 2,6-diidrossiacetofenone.
Tabella 1. Matrici UV-MALDI (Gross J. H., 2006)
| Composto | Sigla | Applicazione a |
|---|---|---|
| Acido nicotinico | NA | Peptidi, proteine |
| Acido picolinico | PA | Oligonucleotidi, DNA |
| Acido 3-idrossipicolinico | HPA, 3-HPA | Oligonucleotidi, DNA |
| Acido 3-Aminopicolinico | 3-APA | Oligonucleotidi, DNA |
| 6-Aza-2-tiotimina | ATT | Oligonucleotidi, DNA |
| Acido 2,5-diidrossibenzoico | DHB | Proteine, oligosaccaridi |
| Miscele a base di DHB | DHB/XY e super-DHB | Proteine, oligosaccaridi |
| 3-Aminochinolina | 3-AQ | Oligosaccaridi |
| acido α-ciano-4-idrossicinnamico | α-CHC, α-CHCA, 4-HCCA, CHCA | Peptidi, proteine minori, triacilgliceroli, numerosi altri composti |
| 4-Cloro-α-ciano-acido cinnamico | ClCCA | Peptidi |
| 3,5-Dimetossi-4-idrossicinnamico | SA | Proteine |
| 2-(4-Idrossifenilazo) acido benzoico | HABA | Peptidi, proteine, glicoproteine, polistirene |
| 2-Mercaptobenzotiazolo | MBT | Peptidi, proteine, polimeri sintetici |
| 5-Cloro-2-mercaptobenzotiazolo | CMBT | Glicopeptidi, fosfopeptidi, e proteine |
| 2,6-Diidrossiacetofenone | DHAP | Glicopeptidi, fosfopeptidi, proteine |
| 2,4,6-Triidrossiacetofenone | THAP | Oligonucleotidi a supporto solido |
| Ditranolo (1,8,9-antracenetriolo) | Nessuno | Polimeri sintetici |
| 9-Nitroantracene | 9-NA | Fullerenes e derivati |
| Benzopirene | Nessuno | Fullerenes e derivati |
| 2-malonitrile | DCTB | Oligomeri, polimeri, dendrimeri, piccole molecole |
Analizzatore TOF (Time of Flight)
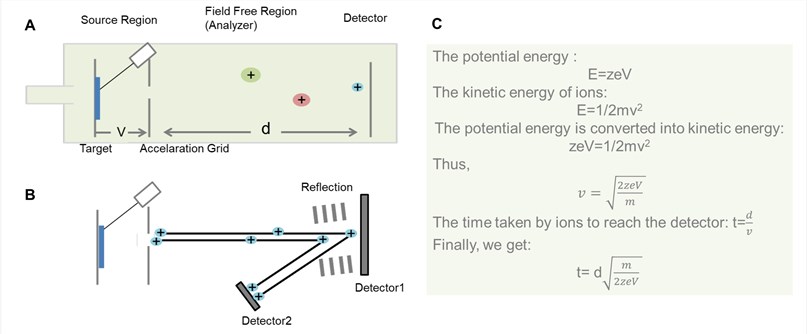
Figura 2. Schema generale dell’analizzatore TOF. (A) Analizzatore TOF di linea; (B) Analizzatore TOF a riflettore; (C) Il processo di derivazione del tempo in cui gli ioni passano attraverso la regione priva di campo nell’analizzatore TOF di linea.
- Il principio della TOF
Come mostrato nella figura 2, il principio di base della TOF è che gli ioni di diverso m/z sono dispersi nel tempo durante il loro volo lungo un percorso di deriva privo di campo di lunghezza nota. A condizione che tutti gli ioni inizino il loro viaggio nello stesso momento o almeno entro un intervallo di tempo sufficientemente breve, quelli più leggeri arriveranno prima al rivelatore rispetto a quelli più pesanti.
- Analizzatore TOF di linea e analizzatore TOF a riflettore
Teoricamente, a tutti gli ioni viene data la stessa energia cinetica iniziale, in modo che dopo la deriva lungo la regione senza campo, gli ioni della stessa m/z al rivelatore al momento. Tuttavia, in pratica, l’impulso non è sentito da tutti gli ioni alla stessa intensità e quindi non tutti gli ioni degli stessi valori m/z raggiungono le loro velocità ideali. Per correggere questo problema, viene spesso applicato un riflettore alla fine della zona di deriva. Il reflectron consiste in una serie di elettrodi ad anello ad alta tensione, che possono respingere gli ioni indietro lungo il tubo di volo di solito con un angolo leggermente spostato.
Ioni di diversa energia cinetica penetrano nel reflectron a diverse profondità prima di essere espulsi dal reflectron nella direzione opposta. Gli ioni più veloci che trasportano più energia cinetica faranno un percorso più lungo di quelli più lenti, e quindi passeranno più tempo all’interno del reflectron rispetto agli ioni più lenti che trasportano meno energia. In questo modo, il rivelatore riceve ioni della stessa massa allo stesso tempo (circa). Quindi, questo design per l’analizzatore di massa TOF ha aumentato la loro risoluzione in modo significativo. Tuttavia, l’analizzatore TOF reflectron non è adatto per gli analiti che non sono abbastanza stabili da sopravvivere al campo elettrico.
Il processo di spettrometria di massa MALDI-TOF
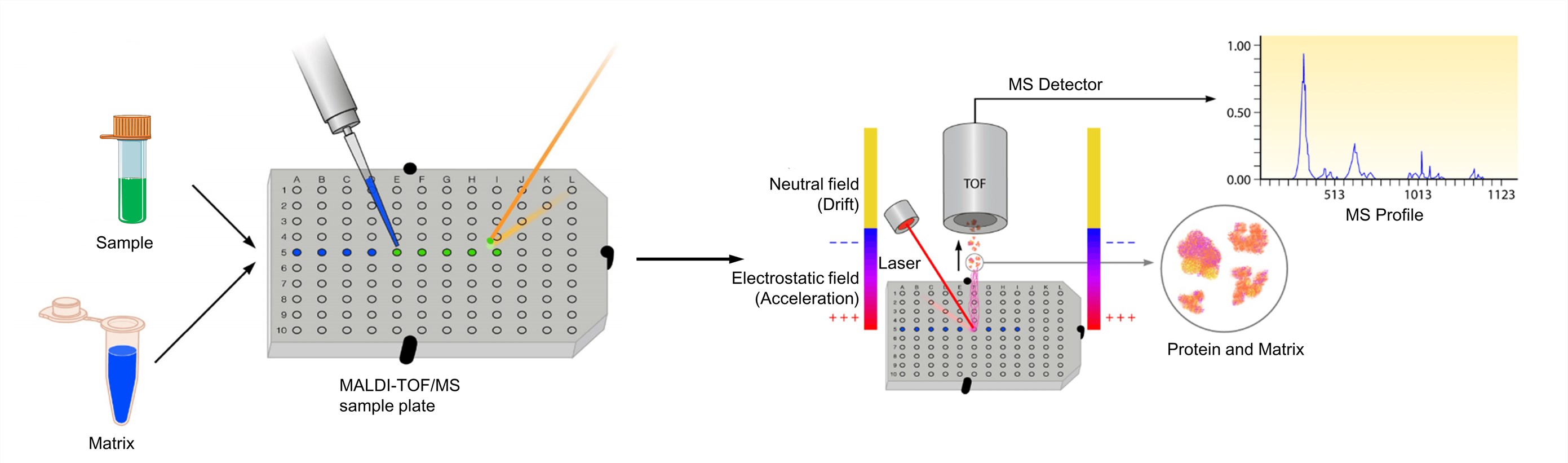
Figura 3. Il processo di spettrometria di massa MALDI-TOF (Clark A. E., et al.; 2013)
L’analita dovrebbe essere solubile ad almeno circa 0,1 mg/ml in qualche solvente. E la matrice viene sciolta per ottenere una soluzione satura o una concentrazione di circa 10 mg/ml. La soluzione dell’analita viene poi mescolata a quella della matrice. Per ottimizzare gli spettri MALDI, il rapporto molare matrice-analita è normalmente regolato in modo da cadere nell’intervallo da 1000: 1 a 100.000: 1. E poi la miscela è poi macchiata su una piastra metallica bersaglio per l’analisi. Dopo l’essiccazione, la miscela di campione e matrice co-cristallizza e forma un deposito solido di campione incorporato nella matrice. La piastra viene successivamente caricata nello strumento MALDI-TOF e analizzata dal software associato al rispettivo sistema. La MALDI porta alla sublimazione e alla ionizzazione sia del campione che della matrice. Questi ioni generati vengono separati a seconda di m/z attraverso un analizzatore TOF, e una rappresentazione spettrale di questi ioni viene generata e analizzata dal software MS, generando un profilo MS.
Applicazione della spettrometria di massa MALDI-TOF
- Determinazione della massa intatta
La determinazione della massa intatta è fondamentale e importante per la caratterizzazione delle proteine, poiché il peso molecolare corretto di una proteina può indicare la struttura intatta. MALDI, una tecnica di ionizzazione morbida, è adatta alle proteine che tendono ad essere fragili e a frammentarsi quando vengono ionizzate con altri metodi di ionizzazione. Le prestazioni di MALDI-TOF MS sono meno influenzate da componenti del buffer, detergenti e contaminanti. Inoltre, permette la determinazione della massa della proteina intatta con una precisione sufficiente (≤ 500 ppm) per la convalida della sequenza. Dopo la digestione delle proteine, la MALDI-TOF MS può essere utilizzata anche per analizzare i peptidi ottenuti per un’ulteriore conferma della sequenza primaria tramite il peptide mass fingerprinting.
- Peptide mass fingerprinting (PMF)
La spettrometria di massa MALDI-TOF ha un funzionamento semplice, una buona precisione di massa, così come un’alta risoluzione e sensibilità. Pertanto, è molto utilizzata nella proteomica per identificare le proteine da miscele semplici con un metodo chiamato peptide mass fingerprinting, che sono spesso utilizzati con l’elettroforesi bidimensionale su gel (2-DE). In questo approccio, i peptidi sono generati dalla digestione delle proteine di interesse con un enzima specifico della sequenza come la tripsina. E poi i peptidi sono analizzati dalla spettrometria di massa MALDI-TOF per ottenere le masse dei peptidi. Le masse sperimentali vengono confrontate con un database contenente le masse teoriche dei peptidi di un dato organismo con la stessa proteasi sequenza-specifica.
- Post source decay (PSD) MALDI-TOF analysis
Gli spettrometri di massa MALDI-TOF dotati di riflettori possono analizzare ioni frammento prodotti da ioni precursori che si decompongono spontaneamente nel volo. Tali ioni sono generalmente indicati come ioni metastabili, e il processo di decomposizione nella regione senza campo tra la sorgente ionica e il riflettore è comunemente indicato come PSD. Gli ioni frammento PSD si formano nella regione senza campo prima di entrare nel riflettore. Gli ioni frammento PSD possono essere separati, raccolti e registrati sul rivelatore cambiando continuamente la tensione del riflettore per formare uno spettro di massa PSD che fornisce informazioni strutturali molto ricche ed efficaci per la struttura primaria di peptidi e proteine. Nello studio di proteomica, alcuni campioni di proteine separati dalla 2DE non possono essere identificati dalla PMF o i risultati dell’identificazione non sono chiari. La funzione di sequenziamento PSD può essere applicata all’identificazione di queste proteine. Usando la spettroscopia PSD, combinata con una ricerca nel database, le proteine possono essere identificate rapidamente e con alta specificità.
- Analisi degli oligonucleotidi
Con lo sviluppo delle tecniche di biologia molecolare e delle tecnologie dei farmaci di acido nucleico antisenso, sempre più frammenti di oligonucleotidi sono stati sintetizzati per essere usati come primer, sonde e farmaci antisenso. È assolutamente necessario rilevare rapidamente questi frammenti per determinare se la sintesi è completa e se la sequenza sintetizzata è corretta. La spettrometria di massa, compresa la MALDI-TOF-MS, è di gran lunga il mezzo migliore per farlo. L’analisi degli oligonucleotidi con MALDI-TOF-MS è semplice, rapida, accurata e sensibile, e può essere utilizzata per determinare la sequenza completa dell’oligonucleotide.
- MALDI imaging
Il MALDI-TOF può essere utilizzato nel profiling e nell’imaging delle proteine direttamente da sezioni sottili di tessuto, noto come spettrometria di massa MALDI imaging (MALDI-IMS). Fornisce informazioni specifiche sulla composizione molecolare locale, l’abbondanza relativa e la distribuzione spaziale di peptidi e proteine nella sezione analizzata. MALDI-IMS può analizzare più composti sconosciuti in sezioni di tessuto biologico simultaneamente attraverso una singola misurazione che può ottenere l’imaging molecolare del tessuto mantenendo l’integrità delle cellule e delle molecole nei tessuti.
La spettrometria di massa MALDI-TOF può analizzare un’ampia varietà di biomolecole, come peptidi, proteine, carboidrati, oligonucleotidi e così via. A causa del fatto che gli ioni formati hanno una bassa energia interna, un grande vantaggio della MALDI-TOF è che il processo di soft-ionizzazione permette di osservare le molecole ionizzate con poca frammentazione degli analiti, permettendo di identificare gli ioni molecolari degli analiti, anche all’interno delle miscele. Ed è facile da usare e mantenere con una rapida acquisizione dei dati. La scelta della matrice appropriata è importante per il successo della spettrometria di massa MALDI-TOF.
A Creative Proteomics, possiamo fornire vari servizi basati sulle nostre piattaforme avanzate di spettrometria di massa MALDI-TOF, tra cui:
- Determinazione della massa molecolare
- Peptide Mass Fingerprinting (PMF)
- Imaging Mass Spectrometry (IMS)
1. Gross J H. Spettrometria di massa: un libro di testo. Springer Science & Business Media, 2006.
2. Boesl U. Time – of – flight mass spectrometry: Introduzione alle basi. Recensioni di spettrometria di massa, 2017, 36(1): 86-109.
3. Guerrera I C, Kleiner O. Applicazione della spettrometria di massa nella proteomica. Bioscience Reports, 2005, 25(1-2): 71-93.
4. Fuchs B, Schiller J. Applicazione della spettrometria di massa MALD – TOF nella lipidomica. European Journal of Lipid Science and Technology, 2009, 111(1): 83-98.
5. Duncan M W, Roder H, Hunsucker S W. Spettrometria di massa quantitativa a desorbimento/ionizzazione laser assistita da matrice. Briefings in genomica funzionale e proteomica, 2008, 7 (5): 355-370.
6. Kenny D J, Brown J M, Palmer M E, et al. Un approccio parallelo all’analisi MALDI-TOF post decadimento sorgente. Journal of the American Society for Mass Spectrometry, 2006, 17(1): 60-66.