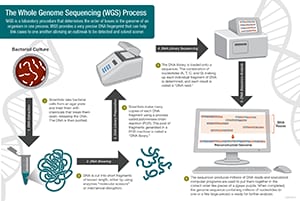
WGSのワークフローです。
全ゲノム配列決定(WGS)とは
生物(バクテリア、ウイルス、ジャガイモ、ヒト)のゲノム(遺伝物質)はDNAで構成されています。 各生物は、塩基(A、T、C、G)で構成される固有のDNA配列を持っています。 ある生物の塩基配列がわかれば、その生物に固有のDNAの指紋(パターン)がわかったことになります。 塩基の順番を決めることを「塩基配列決定」といいます。
全ゲノム配列決定の仕組み
科学者は、以下の4つの主要なステップを踏んで全ゲノム配列決定を行っています。 科学者はまず、分子はさみを使って、何百万もの塩基で構成されているDNAを切断します。

全ゲノム配列は病気の検出をどのように変えるか?
全ゲノムシーケンスは、PulseNetが使用している現在の標準的な技術であるパルスフィールドゲル電気泳動(PFGE)よりも、アウトブレイクを特定するためのより詳細で正確なデータを提供します。 PFGEパターンに現れる15~30のバンドを使って細菌ゲノムを比較することしかできなかったのが、今では何百万もの塩基を比較することができます。 これは、本の章の数(PFGE)だけではなく、本の中のすべての単語(WGS)を比較して、本が同じか違うかを調べるようなものです。 全ゲノム配列解析を行うことで、PFGEでは異なると思われた細菌が、実は同じ由来であることがわかりました。
全ゲノム塩基配列は、たった1回の検査で細菌に関する高度な情報を迅速かつ安価に入手できる方法です。 現在、細菌の特徴を完全に把握するためのプロセスでは、2人以上の科学者がPFGEを含む4つ以上の別々の検査を行う必要があります。
PulseNetは、次世代シーケンシング(NGS)技術の検証や、データ解析に必要なツールの開発、評価、導入を積極的に行っています。
Meeting the challenges of whole genome sequencing.
2013年、CDCは致死性細菌であるリステリア菌によるアウトブレイクを検出するために、全ゲノム配列の使用を開始しました。 それ以来、この方法によって科学者たちは以下のことができるようになりました。
- より多くのリステリア菌の集団感染を検出する
- より多くのリステリア菌の集団感染を、まだ小さいうちに解決する
- 病気の患者を、可能性の高い食品源に結びつける
- リステリア菌の新しい食品源を特定する。
Listeria Whole Genome Sequencing Project が食品由来のアウトブレイクの検出と調査をどのように改善したかについては、こちらをご覧ください。
CDCは州の研究所での全ゲノム配列の使用を急速に拡大しており、科学者たちはまもなく、カンピロバクター、志賀毒素産生性大腸菌(STEC)、サルモネラなど、他の食中毒病原体のアウトブレイク調査に全ゲノム配列を使用し始めるでしょう。
CDCのAMD(Advanced Molecular Detection)イニシアチブと食品安全プログラムとの連携により、PulseNetは、全ゲノムシーケンスへの移行をサポートする体制を整えています。これには、シーケンスを実施する公衆衛生微生物学者のトレーニング、シーケンス用品の購入、データ解析システムの更新などが含まれます。 これらの活動は、公衆衛生の研究室で全ゲノムシーケンスを開始し、食中毒の発生や食品由来の感染症や抗生物質耐性の傾向に関する監視を改善するために不可欠です。
全ゲノムシーケンスの使用が拡大するにつれ、CDCの全国的な監視システムと研究室のインフラは、変化する技術に対応しなければなりません。
全ゲノム配列の使用が拡大するにつれ、CDCの全国的な監視システムと研究所のインフラは、技術の変化に対応しなければなりません。 また、食品由来のアウトブレイクを迅速に解決し、防止するために必要な証拠を確保することができます。