Massenspektrometrie ist eine analytische Technik, bei der Proben in geladene Moleküle ionisiert werden und das Verhältnis ihrer Masse-zu-Ladung (m/z) gemessen werden kann. Bei der MALDI-TOF-Massenspektrometrie ist die Ionenquelle die Matrix-unterstützte Laser-Desorption/Ionisation (MALDI) und der Massenanalysator ist der Time-of-Flight (TOF)-Analysator.
MALDI
MALDI ist eine weiche Ionisation, bei der ein Laser auf eine Matrix kleiner Moleküle trifft, um die Analytmoleküle in die Gasphase zu bringen, ohne sie zu fragmentieren oder zu zersetzen. Einige Biomoleküle sind zu groß und können sich beim Erhitzen zersetzen, und herkömmliche Techniken würden Makromoleküle fragmentieren oder zerstören. MALDI ist geeignet, um Biomoleküle wie Peptide, Lipide, Saccharide oder andere organische Makromoleküle zu analysieren.
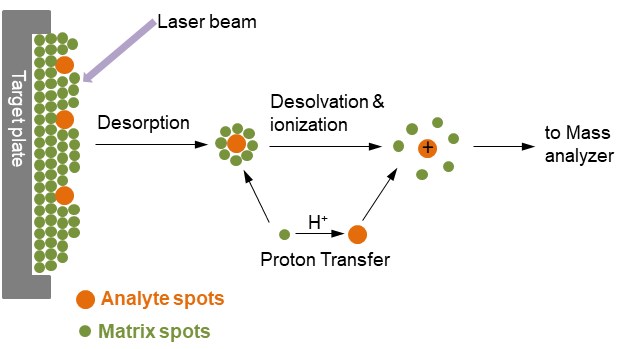
Abbildung 1. Ionisierung von Analyten mittels MALDI
- Das Prinzip von MALDI
In Abbildung 1 ist der Analyt in einen sehr großen Überschuss einer Matrixverbindung eingebettet, die auf einer festen Oberfläche, dem sogenannten Target, aufgebracht ist, das in der Regel aus einem leitenden Metall besteht und Spots für mehrere verschiedene Proben aufweist. Nach einem sehr kurzen Laserpuls wird der bestrahlte Spot schnell erhitzt und zu Schwingungen angeregt. Die Matrixmoleküle, die von der Oberfläche der Probe energetisch abgetragen werden, absorbieren die Laserenergie und tragen die Analytmoleküle mit in die Gasphase. Während des Ablationsprozesses werden die Analytmoleküle normalerweise ionisiert, indem sie mit den nahegelegenen Matrixmolekülen protoniert oder deprotoniert werden. Das gängigste MALDI-Ionisierungsformat ist, dass die Analytmoleküle eine einzelne positive Ladung tragen.
- Gängige Lasertypen für MALDI
Laser sowohl mit ultravioletten (UV) als auch mit infraroten (IR) Wellenlängen sind im Einsatz, aber UV-Laser sind bei weitem die wichtigsten Lichtquellen für analytisches MALDI. Unter diesen dienen Stickstofflaser und frequenzverdreifachte oder -vervierfachte Nd:Yag-Laser oft für die Mehrzahl der Anwendungen. IR-MALDI wird von Er:Yag-Lasern dominiert, während TEA-CO2-Laser selten verwendet werden.
- Gebräuchliche MALDI-Matrixsubstanz
Es wird angenommen, dass die erste Funktion der Matrix im Wesentlichen darin besteht, die Analytmoleküle zu verdünnen und voneinander zu isolieren. Dies geschieht bei der Verdampfung des Lösungsmittels und der damit einhergehenden Bildung eines Festkörpers. Bei der Laserbestrahlung fungiert sie dann als Vermittler für die Energieabsorption. Die Wahl der richtigen Matrix ist der Schlüssel zum Erfolg bei MALDI. Im Allgemeinen funktionieren hochpolare Analyten besser mit hochpolaren Matrizen, und unpolare Analyten werden vorzugsweise mit unpolaren Matrizen kombiniert. Wie in Tabelle 1 dargestellt, wurden verschiedene Matrizen gesucht und häufig verwendet. Die derzeit am häufigsten verwendeten Matrices sind α-Cyano-4-hydroxyzimtsäure, 2,5-Dihydroxybenzoesäure, 3,5-Dimethoxy-4-hydroxyzimtsäure und 2,6-Dihydroxyacetophenon.
Tabelle 1. UV-MALDI-Matrizen (Gross J. H., 2006)
| Verbindung | Akronym | Anwendung zu |
|---|---|---|
| Nikotinsäure | NA | Peptide, Proteine |
| Picolinsäure | PA | Oligonukleotide, DNA |
| 3-Hydroxypicolinsäure | HPA, 3-HPA | Oligonukleotide, DNA |
| 3-Aminopicolinsäure | 3-APA | Oligonukleotide, DNA |
| 6-Aza-2-thiothymine | ATT | Oligonukleotide, DNA |
| 2,5-Dihydroxybenzoesäure | DHB | Proteine, Oligosaccharide |
| DHB-basierte Mischungen | DHB/XY und Super-DHB | Proteine, Oligosaccharide |
| 3-Aminochinolin | 3-AQ | Oligosaccharide |
| α-Cyano-4-hydroxyzimtsäure | α-CHC, α-CHCA, 4-HCCA, CHCA | Peptide, kleinere Proteine, Triacylglycerine, zahlreiche andere Verbindungen |
| 4-Chlor-α-cyano-Zimtsäure | ClCCA | Peptide |
| 3,5-Dimethoxy-4-hydroxyzimtsäure | SA | Proteine |
| 2-(4-Hydroxyphenylazo)benzoesäure | HABA | Peptide, Proteine, Glykoproteine, Polystyrol |
| 2-Mercaptobenzothiazol | MBT | Peptide, Proteine, synthetische Polymere |
| 5-Chlor-2-mercaptobenzothiazol | CMBT | Glycopeptide, Phosphopeptide, und Proteine |
| 2,6-Dihydroxyacetophenon | DHAP | Glykopeptide, Phosphopeptide, Proteine |
| 2,4,6-Trihydroxyacetophenon | THAP | Festkörpergestützte Oligonukleotide |
| Dithranol (1,8,9-Anthracentriol) | Keine | Synthetische Polymere |
| 9-Nitroanthracen | 9-NA | Fullerene und Derivate |
| Benzopyren | Keine | Fullerene und Derivate |
| 2-Malonitril | DCTB | Oligomere, Polymere, Dendrimere, kleine Moleküle |
Time of Flight (TOF) Analysator
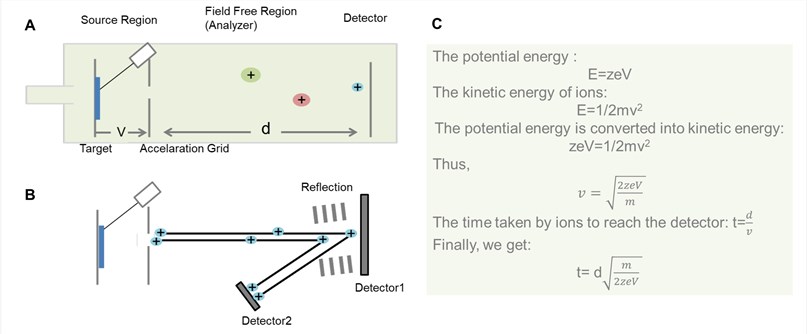
Abbildung 2. Allgemeines Schema für TOF-Analysator. (A) Liner-TOF-Analysator; (B) Reflektor-TOF-Analysator; (C) Der Ableitungsprozess der Zeit, die Ionen durch den feldfreien Bereich im Liner-TOF-Analysator laufen.
- Das Prinzip der TOF
Wie in Abbildung 2 dargestellt, ist das Grundprinzip der TOF, dass Ionen unterschiedlicher m/z während ihres Fluges entlang einer feldfreien Driftstrecke bekannter Länge zeitlich gestreut werden. Unter der Voraussetzung, dass alle Ionen ihre Reise zur gleichen Zeit oder zumindest innerhalb eines ausreichend kurzen Zeitintervalls beginnen, kommen die leichteren früher am Detektor an als die schwereren.
- Liner TOF-Analysator und Reflektron TOF-Analysator
Theoretisch erhalten alle Ionen die gleiche kinetische Anfangsenergie, so dass nach der Drift entlang der feldfreien Region die Ionen der gleichen m/z am Detektor zur gleichen Zeit ankommen. In der Praxis wird der Impuls jedoch nicht von allen Ionen mit der gleichen Intensität empfunden, so dass nicht alle Ionen der gleichen m/z-Werte ihre ideale Geschwindigkeit erreichen. Um dieses Problem zu korrigieren, wird oft eine Reflexion am Ende der Driftzone angebracht. Das Reflektron besteht aus einer Reihe von Ringelektroden mit hoher Spannung, die die Ionen entlang des Flugrohrs meist in einem leicht versetzten Winkel zurückstoßen können.
Ionen unterschiedlicher kinetischer Energie durchdringen das Reflektron unterschiedlich tief, bevor sie in die entgegengesetzte Richtung aus dem Reflektron ausgestoßen werden. Schnellere Ionen mit höherer kinetischer Energie legen einen längeren Weg zurück als langsamere und verbringen daher mehr Zeit im Reflektron als langsamere Ionen mit geringerer Energie. Auf diese Weise empfängt der Detektor Ionen der gleichen Masse zu (etwa) der gleichen Zeit. Dadurch hat dieses Design für TOF-Massenanalysatoren deren Auflösung deutlich erhöht. Allerdings ist der Reflektron-TOF-Analysator nicht für Analyten geeignet, die nicht stabil genug sind, um das elektrische Feld zu überstehen.
Der Prozess der MALDI-TOF-Massenspektrometrie
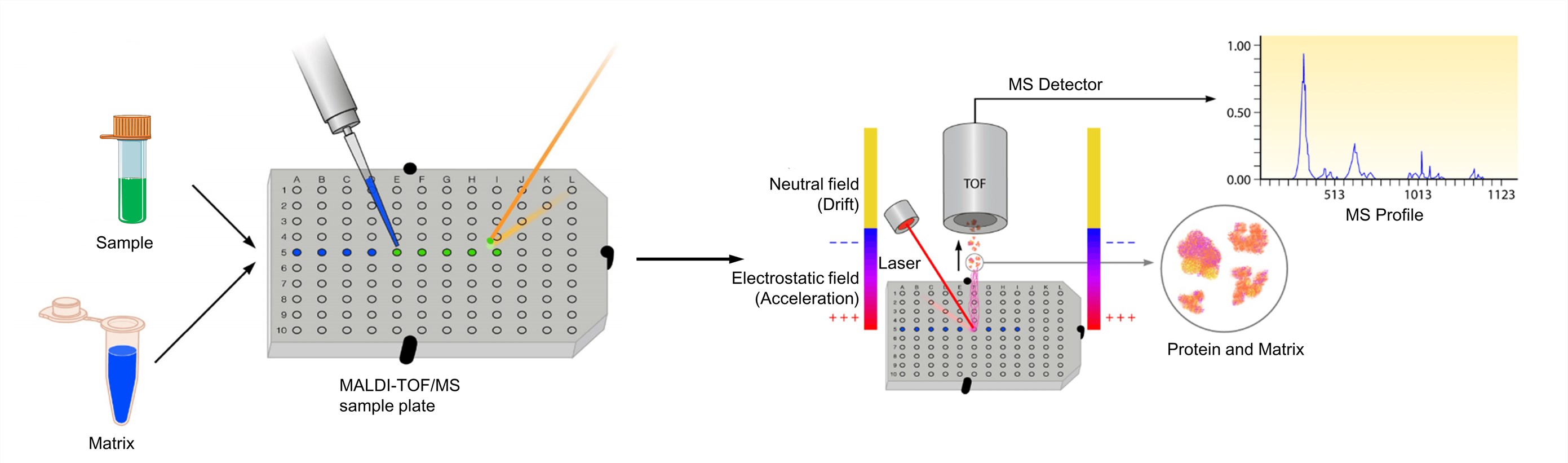
Abbildung 3. Der Prozess der MALDI-TOF-Massenspektrometrie (Clark A. E., et al.; 2013)
Der Analyt sollte zu mindestens etwa 0,1 mg/ml in einem Lösungsmittel löslich sein. Und die Matrix wird gelöst, um entweder eine gesättigte Lösung oder eine Konzentration von etwa 10 mg/ml zu erhalten. Die Lösung des Analyten wird dann derjenigen der Matrix zugemischt. Für optimierte MALDI-Spektren wird das molare Matrix-zu-Analyt-Verhältnis normalerweise so eingestellt, dass es im Bereich von 1000: 1 bis 100.000: 1 liegt. Anschließend wird das Gemisch zur Analyse auf eine Metalltargetplatte getupft. Nach dem Trocknen kristallisiert das Gemisch aus Probe und Matrix zusammen und bildet eine feste, in die Matrix eingebettete Ablagerung der Probe. Die Platte wird anschließend in das MALDI-TOF-Gerät geladen und von der mit dem jeweiligen System verbundenen Software analysiert. Das MALDI führt zur Sublimation und Ionisierung sowohl der Probe als auch der Matrix. Diese erzeugten Ionen werden in Abhängigkeit von m/z durch einen TOF-Analysator getrennt, und eine spektrale Darstellung dieser Ionen wird erzeugt und von der MS-Software analysiert, wodurch ein MS-Profil entsteht.
Anwendung der MALDI-TOF-Massenspektrometrie
- Intaktmassenbestimmung
Die Intaktmassenbestimmung ist grundlegend und wichtig für die Proteincharakterisierung, da das richtige Molekulargewicht eines Proteins auf die intakte Struktur hinweisen kann. MALDI, eine weiche Ionisierungstechnik, eignet sich für Proteine, die dazu neigen, fragil zu sein und zu fragmentieren, wenn sie mit anderen Ionisierungsmethoden ionisiert werden. Die Leistung von MALDI-TOF MS wird durch Pufferkomponenten, Detergenzien und Verunreinigungen weniger beeinträchtigt. Darüber hinaus ermöglicht sie die Bestimmung der Masse intakter Proteine mit ausreichender Genauigkeit (≤ 500 ppm) für die Sequenzvalidierung. Nach dem Proteinverdau kann MALDI-TOF MS auch zur Analyse der erhaltenen Peptide für eine weitere primäre Sequenzbestätigung durch Peptid-Massen-Fingerprinting verwendet werden.
- Peptid-Massen-Fingerprinting (PMF)
Die MALDI-TOF-Massenspektrometrie zeichnet sich durch einfache Bedienung, gute Massengenauigkeit sowie hohe Auflösung und Empfindlichkeit aus. Daher ist sie in der Proteomik weit verbreitet, um Proteine aus einfachen Gemischen durch eine Methode namens Peptid-Massen-Fingerprinting zu identifizieren, die oft mit zweidimensionaler Gelelektrophorese (2-DE) verwendet wird. Bei diesem Ansatz werden Peptide durch den Verdau von Proteinen von Interesse mit einem sequenzspezifischen Enzym wie Trypsin erzeugt. Anschließend werden die Peptide mittels MALDI-TOF-Massenspektrometrie analysiert, um die Peptidmassen zu erhalten. Die experimentellen Massen werden mit einer Datenbank verglichen, die theoretische Peptidmassen aus einem bestimmten Organismus mit der gleichen sequenzspezifischen Protease enthält.
- Post Source Decay (PSD) MALDI-TOF-Analyse
MALDI-TOF-Massenspektrometer, die mit Reflektronen ausgestattet sind, können Fragment-Ionen analysieren, die von Vorläufer-Ionen erzeugt werden, die sich spontan im Flug zersetzen. Solche Ionen werden allgemein als metastabile Ionen bezeichnet, und der Prozess der Zersetzung im feldfreien Bereich zwischen der Ionenquelle und dem Reflektron wird allgemein als PSD bezeichnet. PSD-Fragment-Ionen werden innerhalb des feldfreien Bereichs gebildet, bevor sie in das Reflektron eintreten. PSD-Fragment-Ionen können getrennt, gesammelt und auf dem Detektor aufgezeichnet werden, indem die Reflektorspannung kontinuierlich geändert wird, um ein PSD-Massenspektrum zu bilden, das sehr reiche und effektive Strukturinformationen für die Primärstruktur von Peptiden und Proteinen liefert. In der Proteomics-Studie können einige 2DE-getrennte Proteinproben nicht durch PMF identifiziert werden oder die Ergebnisse der Identifizierung sind nicht eindeutig. Die PSD-Sequenzierungsfunktion kann für die Identifizierung dieser Proteine eingesetzt werden. Mit Hilfe der PSD-Spektroskopie, kombiniert mit einer Datenbanksuche, können Proteine schnell und mit hoher Spezifität identifiziert werden.
- Oligonukleotid-Analyse
Mit der Entwicklung von molekularbiologischen Techniken und Antisense-Nukleinsäure-Medikamenten-Technologien wurden mehr und mehr Oligonukleotid-Fragmente synthetisiert, die als Primer, Sonden und Antisense-Medikamente verwendet werden. Es ist durchaus notwendig, diese Fragmente schnell zu erkennen, um festzustellen, ob die Synthese vollständig ist und ob die synthetisierte Sequenz korrekt ist. Die Massenspektrometrie, einschließlich MALDI-TOF-MS, ist bei weitem das beste Mittel, dies zu tun. Die Oligonukleotid-Analyse mit MALDI-TOF-MS war einfach, schnell, genau und empfindlich und kann zur Bestimmung der vollständigen Oligonukleotid-Sequenz verwendet werden.
- MALDI-Imaging
Das MALDI-TOF kann zur Profilierung und zum Imaging von Proteinen direkt aus dünnen Gewebeschnitten verwendet werden, was als MALDI-Imaging-Massenspektrometrie (MALDI-IMS) bekannt ist. Sie liefert spezifische Informationen über die lokale molekulare Zusammensetzung, relative Häufigkeit und räumliche Verteilung von Peptiden und Proteinen im analysierten Schnitt. MALDI-IMS kann mehrere unbekannte Verbindungen in biologischen Gewebeschnitten gleichzeitig durch eine einzige Messung analysieren, die ein Molekül-Imaging des Gewebes erhalten kann, während die Integrität der Zellen und Moleküle in den Geweben erhalten bleibt.
MALDI-TOF-Massenspektrometrie kann eine Vielzahl von Biomolekülen analysieren, wie Peptide, Proteine, Kohlenhydrate, Oligonukleotide und so weiter. Aufgrund der Tatsache, dass die gebildeten Ionen eine niedrige innere Energie haben, ist ein großer Vorteil von MALDI-TOF, dass der Prozess der Soft-Ionisierung die Beobachtung der ionisierten Moleküle mit geringer Fragmentierung der Analyten ermöglicht, so dass die molekularen Ionen der Analyten identifiziert werden können, sogar in Mischungen. Außerdem ist es einfach zu bedienen und zu warten mit schneller Datenerfassung. Wichtig für eine erfolgreiche MALDI-TOF-Massenspektrometrie ist die Wahl der geeigneten Matrixsubstanz.
Bei Creative Proteomics können wir verschiedene Dienstleistungen auf der Basis unserer fortschrittlichen MALDI-TOF-Massenspektrometrie-Plattformen anbieten, darunter:
- Molekularmassenbestimmung
- Peptid-Massen-Fingerprinting (PMF)
- Imaging-Massenspektrometrie (IMS)
1. Gross J H. Massenspektrometrie: ein Lehrbuch. Springer Science & Business Media, 2006.
2. Boesl U. Time – of – flight mass spectrometry: Einführung in die Grundlagen. Mass spectrometry reviews, 2017, 36(1): 86-109.
3. Guerrera I C, Kleiner O. Application of mass spectrometry in proteomics. Bioscience Reports, 2005, 25(1-2): 71-93.
4. Fuchs B, Schiller J. Application of MALD – TOF mass spectrometry in lipidomics. European Journal of Lipid Science and Technology, 2009, 111(1): 83-98.
5. Duncan M W, Roder H, Hunsucker S W. Quantitative matrix-assisted laser desorption/ionization mass spectrometry. Briefings in Functional Genomics and Proteomics, 2008, 7(5): 355-370.
6. Kenny D J, Brown J M, Palmer M E, et al. A parallel approach to post source decay MALDI-TOF analysis. Journal of the American Society for Mass Spectrometry, 2006, 17(1): 60-66.