Espectrometria de massa é uma técnica analítica na qual as amostras são ionizadas em moléculas carregadas e a proporção da sua massa/carga (m/z) pode ser medida. Na espectrometria de massa MALDI-TOF, a fonte de iões é a dessorção/ionização laser assistida por matriz (MALDI), e o analisador de massa é o analisador de tempo de voo (TOF).
MALDI
MALDI é uma ionização suave que envolve um laser que atinge uma matriz de pequenas moléculas para fazer com que as moléculas analisadas entrem na fase gasosa sem as fragmentar ou decompor. Algumas biomoléculas são demasiado grandes e podem decompor-se quando aquecidas, e as técnicas tradicionais irão fragmentar ou destruir as macromoléculas. MALDI é apropriado para analisar biomoléculas como peptídeos, lípidos, sacarídeos, ou outras macromoléculas orgânicas.
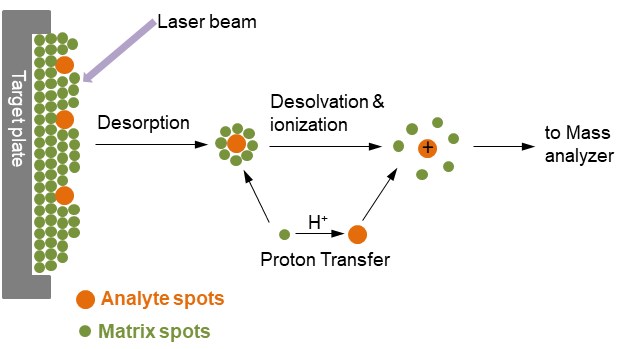
Figure 1. Ionização de analitos por MALDI
- O princípio de MALDI
Na Figura 1, a substância a analisar está embutida num excesso muito grande de um composto matricial depositado sobre uma superfície sólida chamada alvo, normalmente feita de um metal condutor e com manchas para várias amostras diferentes a serem aplicadas. Após um impulso laser muito breve, a mancha irradiada é rapidamente aquecida e torna-se vibrantemente excitada. As moléculas da matriz são energeticamente ablacionadas da superfície da amostra, absorvem a energia do laser e transportam as moléculas da substância a analisar também para a fase gasosa. Durante o processo de ablação, as moléculas da substância a analisar são normalmente ionizadas por serem protonadas ou desprotonadas com as moléculas de matriz próximas. O formato mais comum de ionização do MALDI é que as moléculas a analisar transportem uma única carga positiva.
- li>Tipos de laser comummente utilizados no MALDI
Lasers tanto de comprimento de onda ultravioleta (UV) como infravermelho (IR) estão em uso, mas os lasers UV são de longe as fontes de luz mais importantes no MALDI analítico. Entre estas, os lasers de azoto e os lasers de frequência triplicada ou quadruplicada Nd: Os lasers Yag servem frequentemente para a maioria das aplicações. O IR-MALDI é dominado pelos lasers Er:Yag enquanto que os lasers TEA-CO2 são raramente utilizados.
- li>Substância da matriz de MALDI comummente utilizada
Acredita-se que a primeira função da matriz é essencialmente diluir e isolar as moléculas de análise umas das outras. Isto ocorre durante a evaporação do solvente e a formação concomitante de uma solução sólida. Depois, após irradiação laser, funciona como um mediador para a absorção de energia. A escolha da matriz certa é a chave para o sucesso no MALDI. Em geral, os analitos altamente polares funcionam melhor com matrizes altamente polares, e os analitos não polares são de preferência combinados com matrizes não polares. Como se mostra no Quadro 1, têm sido procuradas e amplamente utilizadas diferentes matrizes. Actualmente, as matrizes mais utilizadas são α-cyano-4-hydroxycinnamic acid, 2,5-dihydroxybenzoic acid, 3,5-dimethoxy-4-hydroxycinnamic acid, e 2,6-dihydroxyacetophenone.
Table 1. Matrizes UV-MALDI (Gross J. H.., 2006)
| Complexo | Acrónimo | Aplicação to |
|---|---|---|
| Ácido nicotínico | NA | Peptides, proteins |
| Ácido picolinico | PA | Oligonucleótidos, DNA |
| ácido 3-hidroxipicolinico | HPA, 3-HPA | Oligonucleótidos, DNA |
| 3-Acido mineralopicolinico | 3-APA | Oligonucleótidos, DNA |
| 6-Aza-2-thiothymine | ATT | Oligonucleótidos, DNA |
| 2,5-Dihydroxybenzoic acid | DHB | Proteínas, oligossacarídeos |
| misturas baseadas em DHB | DHB/XY e super-DHB | Proteínas, oligossacarídeos |
| 3-Aminoquinolina | 3-AQ | Oligosacáridos |
| α-Cyano-4-hydroxycinnamic acid | α-CHC, α-CHCA, 4-HCCA, CHCA | Peptídeos, proteínas mais pequenas, triactilgliceróis, numerosos outros compostos |
| 4-Cloro-α-cyano-cinnamic acid | ClCCA | Peptides |
| 3,5-Dimethoxy-4-hydroxycinnamic acid | SA | Proteínas |
| 2-(4-Hydroxyphenylazo) benzoic acid | HABA | Peptides, proteínas, glicoproteínas, poliestireno |
| 2-Mercaptobenzothiazole | MBT | Peptides, proteínas, polímeros sintéticos |
| 5-Chloro-2-mercaptobenzothiazole | CMBT | Glycopeptides, fosfopeptídeos, e proteínas |
| 2,6-Dihydroxyacetophenone | DHAP | Glycopeptides, phosphopeptides, proteínas |
| 2,4,6-Trihidroxiacetofenona | THAP | Oligonucleótidos suportados por sólidos |
| Dithranol (1,8,9-anthracenetriol) | Nenhum | Polímeros sintéticos |
| 9-Nitroanthracene | 9-NA | Fullerenes e derivados |
| Benzopyrene | Nenhum | Fullerenes e derivados |
| 2-malonitrilo | DCTB | Oligómeros, polímeros, dendrimers, pequenas moléculas |
Analisador de Tempo de Voo (TOF)
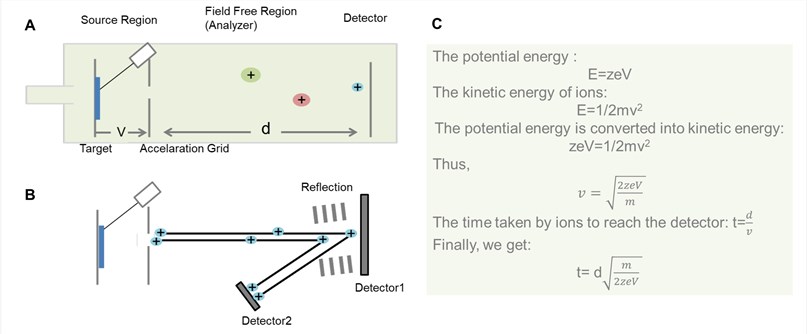
Figure 2. Esquema geral do analisador TOF. (A) Analisador TOF de revestimento; (B) Analisador TOF reflector; (C) O processo de derivação do tempo que os iões passam através da região livre de campo no analisador TOF de revestimento.
- O princípio de TOF
Como mostrado na Figura 2, o princípio básico de TOF é que os iões de diferentes m/z são dispersos no tempo durante o seu voo ao longo de um caminho de deriva livre de campo de comprimento conhecido. Desde que todos os iões comecem a sua viagem ao mesmo tempo ou pelo menos dentro de um intervalo de tempo suficientemente curto, os mais leves chegarão mais cedo ao detector do que os mais pesados.
- li>Liner TOF analisador e reflectron TOF analisador
Teoricamente, todos os iões recebem a mesma energia cinética inicial, de modo que, após a deriva ao longo da região livre de campo, os iões do mesmo m/z no detector na altura. Contudo, na prática, o pulso não é sentido por todos os iões com a mesma intensidade e por isso nem todos os iões dos mesmos valores m/z atingem as suas velocidades ideais. Para corrigir este problema, aplica-se frequentemente uma reflexão ao fim da zona de deriva. O reflector consiste numa série de eléctrodos de anel com alta voltagem, que podem repelir os iões de volta ao longo do tubo de voo geralmente num ângulo ligeiramente deslocado.
Ions de diferentes energias cinéticas penetram o reflector a diferentes profundidades antes de serem expelidos do reflector na direcção oposta. Os iões mais rápidos transportando mais energia cinética percorrerão um caminho mais longo do que os mais lentos, passando assim mais tempo dentro do reflectrão do que os iões mais lentos transportando menos energia. Desta forma, o detector recebe iões da mesma massa (aproximadamente) ao mesmo tempo. Assim, este desenho do analisador de massa TOF aumentou significativamente a sua resolução. Contudo, o analisador TOF reflectron não é adequado para analitos que não são suficientemente estáveis para sobreviver ao campo eléctrico.
O processo de espectrometria de massa MALDI-TOF
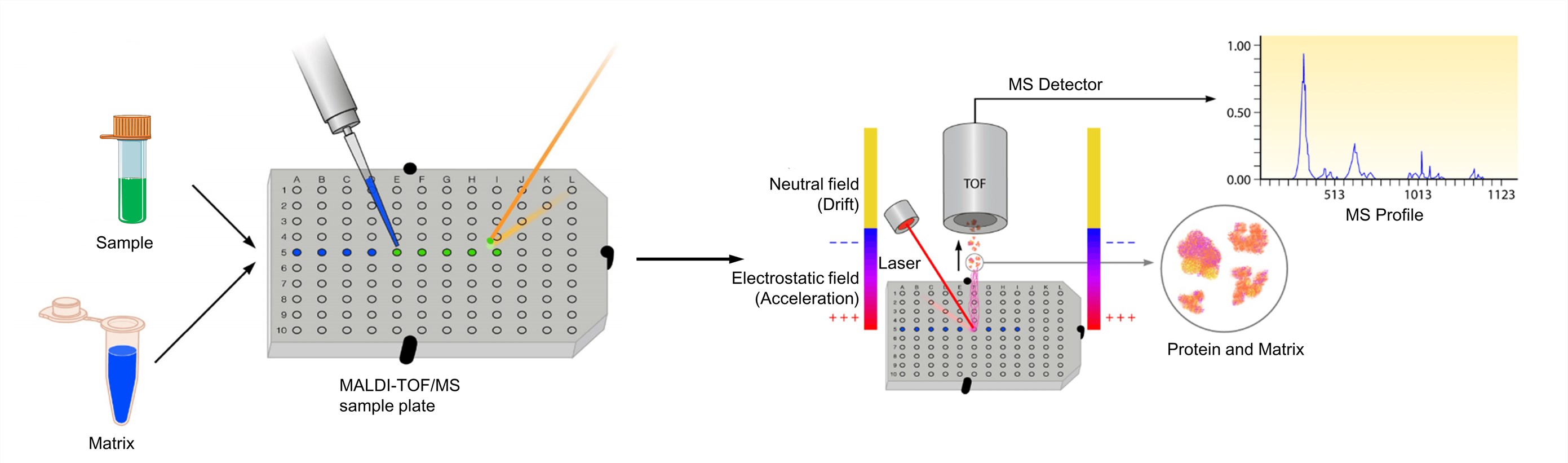
Figure 3. O processo de espectrometria de massa MALDI-TOF (Clark A. E., et al.; 2013)
O analito deve ser solúvel a pelo menos cerca de 0,1 mg/ml em algum solvente. E a matriz é dissolvida para produzir ou uma solução saturada ou uma concentração de cerca de 10 mg/ml. A solução da substância a analisar é então misturada com a da matriz. Para espectros optimizados de MALDI, a razão matriz molar/analito é normalmente ajustada de modo a cair na gama de 1000: 1 a 100.000: 1. E depois a mistura é então manchada para uma placa metálica alvo para análise. Após a secagem, a mistura da amostra e matriz co-cristaliza-se e forma um depósito sólido de amostra incorporado na matriz. A placa é subsequentemente carregada no instrumento MALDI-TOF e analisada por software associado ao respectivo sistema. O MALDI leva à sublimação e ionização tanto da amostra como da matriz. Estes iões gerados são separados dependendo do m/z através de um analisador TOF, e uma representação espectral destes iões é gerada e analisada pelo software MS, gerando um perfil MS.
Aplicação da espectrometria de massa MALDI-TOF
- li>Determinação de massa intacta
A determinação de massa intacta é básica e importante para a caracterização da proteína, devido ao peso molecular correcto de uma proteína pode indicar a estrutura intacta. O MALDI, uma técnica de ionização suave, é adequado para proteínas que tendem a ser frágeis e fragmentadas quando ionizadas por outros métodos de ionização. O desempenho do MALDI-TOF MS é menos afectado por componentes tampão, detergentes, e contaminantes. Além disso, permite a determinação da massa proteica intacta com precisão suficiente (≤ 500 ppm) para validação da sequência. Após a digestão da proteína, MALDI-TOF MS também pode ser utilizado para analisar os peptídeos obtidos para posterior confirmação da sequência primária por impressão digital de massa de peptídeo.
- li>Peptide massa de peptídeo (PMF)
MALDI-TOF espectrometria de massa tem operação simples, boa precisão de massa, bem como alta resolução e sensibilidade. Por conseguinte, tem utilizações generalizadas em proteómica para identificar proteínas de misturas simples por um método chamado impressão digital de massa de peptídeo, que são frequentemente utilizadas com electroforese bidimensional em gel (2-DE). Nesta abordagem, os peptídeos são gerados pela digestão de proteínas de interesse com uma enzima específica da sequência, como a tripsina. E depois os peptídeos são analisados por espectrometria de massa MALDI-TOF para obter as massas dos peptídeos. As massas experimentais são comparadas com uma base de dados contendo massas teóricas de peptídeos de um dado organismo com a mesma protease específica de sequência.
ul>>li>Pós-decadência da fonte (PSD) MALDI-TOF análise
MALDI-TOF espectrómetros de massa equipados com refletores podem analisar iões fragmentos produzidos a partir de iões precursores que se decompõem espontaneamente no voo. Tais iões são geralmente referidos como iões mensuráveis, e o processo de decomposição na região livre de campo entre a fonte de iões e o reflector é geralmente referido como PSD. Os iões fragmentos de PSD são formados dentro da região livre de campo antes de entrarem no reflector. Os iões fragmentos de PSD podem ser separados, recolhidos e registados no detector alterando continuamente a tensão do reflector para formar um espectro de massa PSD que fornece informação estrutural muito rica e eficaz para a estrutura primária de peptídeos e proteínas. No estudo proteómico, algumas amostras de proteínas separadas por 2DE não podem ser identificadas por PMF ou os resultados da identificação não são claros. A função de sequenciação PSD pode ser aplicada à identificação destas proteínas. Usando a espectroscopia PSD, combinada com uma pesquisa na base de dados, as proteínas podem ser identificadas rapidamente e com elevada especificidade.
- li>Análise de oligonucleótidos
Com o desenvolvimento de técnicas de biologia molecular e tecnologias de fármacos antisensores de ácidos nucleicos, cada vez mais fragmentos de oligonucleótidos têm sido sintetizados para serem utilizados como primários, sondas e fármacos antisensores. É inteiramente necessário detectar rapidamente estes fragmentos para determinar se a síntese está completa e se a sequência sintetizada está correcta. A espectrometria de massa, incluindo MALDI-TOF-MS, é de longe o melhor meio para o fazer. A análise de oligonucleótidos utilizando MALDI-TOF-MS foi simples, rápida, precisa e sensível, que pode ser utilizada para determinar a sequência completa de oligonucleótidos.
- li> imagi>MALDI imaging
p> O MALDI-TOF pode ser utilizado na caracterização e imagiologia de proteínas directamente a partir de secções finas de tecido, conhecidas como espectrometria de massa de imagem MALDI (MALDI-IMS). Fornece informação específica sobre a composição molecular local, abundância relativa e distribuição espacial de peptídeos e proteínas na secção analisada. MALDI-IMS pode analisar múltiplos compostos desconhecidos em secções biológicas de tecido simultaneamente através de uma única medição que pode obter a imagem molecular do tecido mantendo a integridade das células e moléculas nos tecidos.
MALDI-TOF espectrometria de massa pode analisar uma grande variedade de biomoléculas, tais como peptídeos, proteínas, hidratos de carbono, oligonucleótido, e assim por diante. Devido ao facto de os iões formados terem baixa energia interna, uma grande vantagem do MALDI-TOF é que o processo de ionização suave permite a observação de moléculas ionizadas com pouca ou nenhuma fragmentação de analitos, permitindo a identificação dos iões moleculares dos analitos, mesmo dentro de misturas. E é fácil de usar e manter com uma rápida aquisição de dados. A escolha da substância matricial apropriada é importante para uma espectrometria de massa MALDI-TOF bem sucedida.
Na Creative Proteomics, podemos fornecer vários serviços baseados nas nossas plataformas avançadas de espectrometria de massa MALDI-TOF, incluindo:
- Determinação de Massa Molecular
- Peptide Mass Fingerprinting (PMF)
- Imaging Mass Spectrometry (IMS)
1. Gross J H. Espectrometria de massa: um livro-texto. Springer Science & Business Media, 2006.
2. Boesl U. Time – of – flight mass spectrometry: Introdução ao básico. Revisões da espectrometria de massa, 2017, 36(1): 86-109.
3. Guerrera I C, Kleiner O. Aplicação da espectrometria de massa em proteómica. Bioscience Reports, 2005, 25(1-2): 71-93.
4. Fuchs B, Schiller J. Aplicação da espectrometria de massa MALD – TOF em lipidómica. European Journal of Lipid Science and Technology, 2009, 111(1): 83-98.
5. Duncan M W, Roder H, Hunsucker S W. Espectrometria de massa por laser de dessorção/ionização quantitativa de matriz. Briefings in functional genomics and proteomics, 2008, 7(5): 355-370.
6. Kenny D J, Brown J M M, Palmer M E, et al. Uma abordagem paralela à análise MALDI-TOF pós-decadência da fonte. Journal of the American Society for Mass Spectrometry, 2006, 17(1): 60-66.
br>