WGS Workflow: Vista maior
Icone pdf de download
O que é a sequenciação do genoma inteiro (WGS)?
O genoma, ou material genético, de um organismo (bactérias, vírus, batata, humano) é constituído por ADN. Cada organismo tem uma sequência única de ADN que é composta de bases (A, T, C, e G). Se conhecer a sequência das bases de um organismo, identificou a sua impressão digital de ADN única, ou padrão. A determinação da ordem das bases é chamada sequenciação. A sequência do genoma inteiro é um procedimento laboratorial que determina a ordem das bases no genoma de um organismo num processo.
Como funciona a sequência do genoma inteiro?
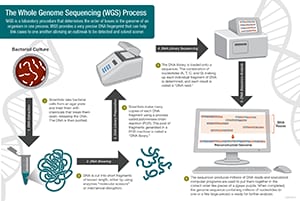
Os cientistas conduzem a sequência do genoma inteiro seguindo estes quatro passos principais:
- tosquia de ADN: Os cientistas começam por utilizar tesouras moleculares para cortar o ADN, que é composto por milhões de bases: A’s, C’s, T’s e G’s, em pedaços suficientemente pequenos para a máquina de sequenciação ler.
- código de barras de ADN: Os cientistas adicionam pequenos pedaços de etiquetas de ADN, ou códigos de barras, para identificar que pedaço de ADN tosquiado pertence a que bactéria. Isto é semelhante a como um código de barras identifica um produto numa mercearia.
- Sequenciação de todo o genoma: O ADN com código de barras de múltiplas bactérias são combinados e colocados em todo o sequenciador genómico. O sequenciador identifica os A’s, C’s, T’s, e G’s, ou bases, que compõem cada sequência bacteriana. O sequenciador utiliza o código de barras para manter o registo de quais as bases que pertencem a quais bactérias.
- Análise de dados: Os cientistas utilizam ferramentas informáticas de análise para comparar sequências bacterianas e identificar diferenças. O número de diferenças pode dizer aos cientistas quão intimamente relacionadas as bactérias estão, e como é provável que façam parte do mesmo surto.

Como é que a sequência do genoma inteiro transformará a detecção de doenças?
A sequenciação de todo o genoma fornece dados mais detalhados e precisos para identificar surtos do que a técnica padrão actual que a PulseNet utiliza, electroforese em gel de campo pulsado (PFGE). Em vez de termos apenas a capacidade de comparar genomas bacterianos usando 15-30 bandas que aparecem num padrão PFGE, temos agora milhões de bases para comparar. Isto é como comparar todas as palavras de um livro (WGS), em vez de apenas o número de capítulos (PFGE), para ver se os livros são os mesmos ou diferentes. Utilizando a sequência do genoma inteiro, descobrimos que algumas bactérias que pareciam ser diferentes utilizando PFGE são na realidade da mesma fonte. Isto ajudou a resolver alguns surtos mais cedo.
A sequenciação de todo o genoma é uma forma rápida e acessível de obter informação de alto nível sobre as bactérias utilizando apenas um teste. Actualmente, o processo de caracterização completa das bactérias requer dois ou mais cientistas para realizar quatro ou mais testes separados, incluindo o PFGE. A WGS irá melhorar consideravelmente a eficiência de como a PulseNet conduz a vigilância.
PulseNet está a validar activamente a tecnologia de sequenciação de próxima geração (NGS), bem como a desenvolver, avaliar e implementar as ferramentas necessárias para analisar os dados.
Conhecendo os desafios da sequenciação de genoma inteiro.
Em 2013, o CDC começou a utilizar a sequenciação de genoma inteiro para detectar surtos causados pela bactéria mortífera Listeria. Desde então, este método tem permitido aos cientistas fazê-lo:
- Detectar mais clusters de doenças de Listeria
- Solver mais surtos de Listeria enquanto ainda são pequenos
- Ligar pacientes doentes a prováveis fontes alimentares
- Identificar novas fontes alimentares de Listeria, tais como maçãs de caramelo e gelados
Saiba mais sobre como o Projecto Listeria Whole Genome Sequencing Project melhorou a detecção e investigação de surtos de origem alimentar.
CDC está a expandir rapidamente o uso de sequenciação do genoma inteiro em laboratórios estatais, e os cientistas começarão em breve a utilizar a sequenciação do genoma inteiro para a investigação de surtos de outros patogéneos de origem alimentar, tais como Campylobacter, E. coli produtora de toxinas Shiga (STEC), e Salmonella. A iniciativa Advanced Molecular Detection (AMD) do CDC financiou parcialmente a expansão do WGS em tempo real para a segurança alimentar.
A PulseNet, através da colaboração com a iniciativa AMD do CDC e o programa de segurança alimentar, está a estabelecer a estrutura para apoiar a mudança para a sequenciação de todo o genoma, incluindo a formação de microbiologistas de saúde pública para realizar a sequenciação, a aquisição de materiais de sequenciação, e a actualização de sistemas de análise de dados. Estas actividades são fundamentais para lançar a sequenciação de todo o genoma em laboratórios de saúde pública e melhorar a vigilância de surtos de doenças de origem alimentar e tendências nas infecções de origem alimentar e resistência aos antibióticos.
A medida que a utilização da sequenciação de todo o genoma se expande, os sistemas nacionais de vigilância do CDC e a infra-estrutura laboratorial devem acompanhar a evolução da tecnologia. Com a modernização, o CDC e os seus parceiros de saúde pública podem continuar a detectar, responder e deter com sucesso as doenças infecciosas. Juntos, podemos assegurar diagnósticos rápidos e menos dispendiosos para indivíduos e as provas necessárias para resolver e prevenir rapidamente surtos de origem alimentar.