WGS Workflow: Larger View
Download pdf icon
Was ist Whole Genome Sequencing (WGS)?
Das Genom, also das genetische Material, eines Organismus (Bakterium, Virus, Kartoffel, Mensch) besteht aus DNA. Jeder Organismus hat eine einzigartige DNA-Sequenz, die aus Basen (A, T, C und G) zusammengesetzt ist. Wenn Sie die Sequenz der Basen in einem Organismus kennen, haben Sie seinen einzigartigen DNA-Fingerabdruck oder sein Muster identifiziert. Die Bestimmung der Reihenfolge der Basen wird als Sequenzierung bezeichnet. Die Ganzgenomsequenzierung ist ein Laborverfahren, das die Reihenfolge der Basen im Genom eines Organismus in einem Prozess bestimmt.
Wie funktioniert die Ganzgenomsequenzierung?
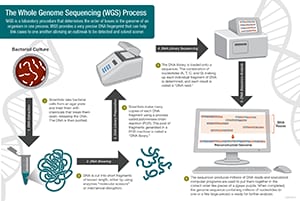
Wissenschaftler führen die Ganzgenomsequenzierung in den folgenden vier Hauptschritten durch:
- DNA-Scheren: Die Wissenschaftler beginnen damit, die DNA, die aus Millionen von Basen besteht, mit einer molekularen Schere zu schneiden: A’s, C’s, T’s und G’s, in Stücke, die klein genug sind, damit die Sequenziermaschine sie lesen kann.
- DNA-Barcoding: Die Wissenschaftler fügen kleine DNA-Stücke, sogenannte Barcodes, hinzu, um zu identifizieren, welches Stück geschorener DNA zu welchem Bakterium gehört. Dies ist vergleichbar mit der Art und Weise, wie ein Strichcode ein Produkt in einem Lebensmittelgeschäft identifiziert.
- Sequenzierung des gesamten Genoms: Die barcodierte DNA von mehreren Bakterien wird kombiniert und in den Whole Genome Sequencer gegeben. Der Sequenzer identifiziert die A’s, C’s, T’s und G’s, oder Basen, aus denen jede bakterielle Sequenz besteht. Der Sequenzer verwendet den Strichcode, um zu verfolgen, welche Basen zu welchen Bakterien gehören.
- Datenanalyse: Wissenschaftler verwenden Computer-Analyse-Tools, um bakterielle Sequenzen zu vergleichen und Unterschiede zu identifizieren. Die Anzahl der Unterschiede kann den Wissenschaftlern sagen, wie eng die Bakterien miteinander verwandt sind und wie wahrscheinlich es ist, dass sie Teil desselben Ausbruchs sind.

Wie wird die Ganzgenomsequenzierung die Erkennung von Krankheiten verändern?
Die Ganzgenomsequenzierung liefert detailliertere und präzisere Daten zur Identifizierung von Krankheitsausbrüchen als die derzeitige Standardtechnik, die PulseNet verwendet, die Pulsed-Field-Gel-Elektrophorese (PFGE). Anstatt nur die Möglichkeit zu haben, bakterielle Genome anhand von 15-30 Banden zu vergleichen, die in einem PFGE-Muster erscheinen, haben wir jetzt Millionen von Basen zum Vergleich. Das ist so, als würde man alle Wörter in einem Buch vergleichen (WGS), anstatt nur die Anzahl der Kapitel (PFGE), um zu sehen, ob die Bücher gleich oder unterschiedlich sind. Mit Hilfe der Ganzgenomsequenzierung haben wir herausgefunden, dass einige Bakterien, die mit PFGE unterschiedlich zu sein schienen, tatsächlich aus derselben Quelle stammen. Dies hat dazu beigetragen, einige Ausbrüche früher aufzuklären.
Die Ganzgenomsequenzierung ist eine schnelle und erschwingliche Möglichkeit, mit nur einem Test hochgradige Informationen über die Bakterien zu erhalten. Derzeit erfordert der Prozess zur vollständigen Charakterisierung von Bakterien zwei oder mehr Wissenschaftler, die vier oder mehr separate Tests einschließlich PFGE durchführen. WGS wird die Effizienz der Überwachung durch PulseNet erheblich verbessern.
PulseNet validiert aktiv die Next-Generation-Sequencing (NGS)-Technologie und entwickelt, evaluiert und implementiert die Tools, die zur Analyse der Daten benötigt werden.
Die Herausforderungen der Ganzgenomsequenzierung meistern.
Im Jahr 2013 begann die CDC mit der Ganzgenomsequenzierung, um Ausbrüche zu erkennen, die durch das tödliche Bakterium Listeria verursacht wurden. Seitdem ermöglicht diese Methode den Wissenschaftlern,:
- Mehr Cluster von Listerien-Erkrankungen erkennen
- Mehr Listerien-Ausbrüche aufklären, solange sie noch klein sind
- Kranke Patienten mit wahrscheinlichen Lebensmittelquellen verknüpfen
- Neue Lebensmittelquellen für Listerien identifizieren, wie Karamelläpfel und Eiscreme
Erfahren Sie mehr darüber, wie das Listeria Whole Genome Sequencing Project die Erkennung und Untersuchung von lebensmittelbedingten Ausbrüchen verbessert hat.
Das CDC weitet den Einsatz der Ganzgenomsequenzierung in staatlichen Labors schnell aus, und Wissenschaftler werden bald damit beginnen, die Ganzgenomsequenzierung für Ausbruchsuntersuchungen anderer lebensmittelbedingter Krankheitserreger wie Campylobacter, Shiga-Toxin-produzierende E. coli (STEC) und Salmonellen einzusetzen. Die CDC-Initiative Advanced Molecular Detection (AMD) finanzierte teilweise die Ausweitung von Echtzeit-WGS für die Lebensmittelsicherheit.
Durch die Zusammenarbeit mit der CDC-Initiative AMD und dem Lebensmittelsicherheitsprogramm baut PulseNet die Struktur auf, um eine Umstellung auf die Ganzgenomsequenzierung zu unterstützen, einschließlich der Schulung von Mikrobiologen des öffentlichen Gesundheitswesens zur Durchführung der Sequenzierung, der Beschaffung von Sequenziermaterial und der Aktualisierung von Systemen für die Datenanalyse. Diese Aktivitäten sind entscheidend für die Einführung der Ganzgenomsequenzierung in den Labors des öffentlichen Gesundheitswesens und für die Verbesserung der Überwachung von lebensmittelbedingten Krankheitsausbrüchen und Trends bei lebensmittelbedingten Infektionen und Antibiotikaresistenzen.
Wenn sich der Einsatz der Ganzgenomsequenzierung ausweitet, müssen die nationalen Überwachungssysteme und die Laborinfrastruktur der CDC mit der sich verändernden Technologie Schritt halten. Mit der Modernisierung können die CDC und ihre Partner im öffentlichen Gesundheitswesen weiterhin erfolgreich Infektionskrankheiten erkennen, darauf reagieren und sie stoppen. Gemeinsam können wir schnelle und weniger kostspielige Diagnosen für Einzelpersonen und die notwendigen Beweise sicherstellen, um lebensmittelbedingte Ausbrüche schnell zu lösen und zu verhindern.