La espectrometría de masas es una técnica analítica en la que las muestras se ionizan en moléculas cargadas y se puede medir la relación entre sus masas y cargas (m/z). En la espectrometría de masas MALDI-TOF, la fuente de iones es la desorción/ionización láser asistida por matriz (MALDI), y el analizador de masas es el analizador de tiempo de vuelo (TOF).
MALDI
La MALDI es una ionización suave que implica que un láser golpee una matriz de moléculas pequeñas para hacer que las moléculas del analito pasen a la fase gaseosa sin fragmentarlas o descomponerlas. Algunas biomoléculas son demasiado grandes y pueden descomponerse al calentarse, y las técnicas tradicionales fragmentarán o destruirán las macromoléculas. MALDI es apropiado para analizar biomoléculas como péptidos, lípidos, sacáridos u otras macromoléculas orgánicas.
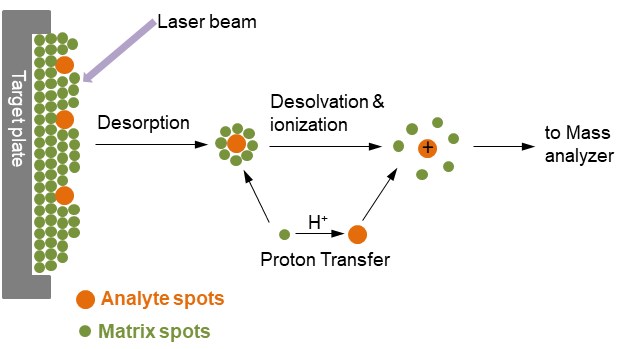
Figura 1. Ionización de analitos por MALDI
- Principio de MALDI
En la figura 1, el analito se incrusta en un exceso muy grande de un compuesto matriz depositado en una superficie sólida llamada blanco, generalmente de un metal conductor y que tiene puntos para varias muestras diferentes que se aplicarán. Tras un pulso láser muy breve, el punto irradiado se calienta rápidamente y se excita vibracionalmente. Las moléculas de la matriz ablacionadas energéticamente en la superficie de la muestra, absorben la energía del láser y llevan las moléculas del analito también a la fase gaseosa. Durante el proceso de ablación, las moléculas de analito suelen ser ionizadas al ser protonadas o desprotonadas con las moléculas de matriz cercanas. El formato de ionización MALDI más común es que las moléculas de analito lleven una sola carga positiva.
- Tipos de láser comúnmente utilizados en MALDI
Se utilizan láseres de longitudes de onda tanto ultravioleta (UV) como infrarroja (IR), pero los láseres UV son, con mucho, las fuentes de luz más importantes en MALDI analítico. Entre ellos, los láseres de nitrógeno y los láseres Nd: Yag de frecuencia triplicada o cuadruplicada suelen servir para la mayoría de las aplicaciones. El IR-MALDI está dominado por los láseres Er:Yag, mientras que los láseres TEA-CO2 se utilizan raramente.
- Sustancia matriz MALDI comúnmente utilizada
Se cree que la primera función de la matriz es esencialmente diluir y aislar las moléculas del analito entre sí. Esto ocurre durante la evaporación del disolvente y la formación concomitante de una solución sólida. A continuación, tras la irradiación láser, funciona como mediador para la absorción de energía. La elección de la matriz adecuada es clave para el éxito en MALDI. En general, los analitos altamente polares funcionan mejor con matrices altamente polares, y los analitos no polares se combinan preferentemente con matrices no polares. Como se muestra en la Tabla 1, se han buscado y utilizado ampliamente diferentes matrices. Actualmente, las matrices más utilizadas son el ácido α-ciano-4-hidroxicinámico, el ácido 2,5-dihidroxibenzoico, el ácido 3,5-dimetoxi-4-hidroxicinámico y la 2,6-dihidroxiacetofenona.
Tabla 1. Matrices UV-MALDI (Gross J. H., 2006)
| Compuesto | Acrónimo | Aplicación a |
|---|---|---|
| Ácido nicotínico | NA | Péptidos, proteínas |
| Ácido picolínico | PA | Oligonucleótidos, ADN |
| Ácido 3-Hidroxipicolínico | HPA, 3-HPA | Oligonucleótidos, ADN |
| Ácido 3-Aminopicolínico | 3-APA | Oligonucleótidos, ADN |
| 6-Aza-2-tiotimina | ATT | Oligonucleótidos, ADN |
| Ácido 2,5-Dihidroxibenzoico | DHB | Proteínas, oligosacáridos | Mezclas a base de DHB | DHB/XY y super-DHB | Proteínas, oligosacáridos | 3-Aminoquinolina | 3-AQ | Oligosacáridos |
| α-Cyano-4-hydroxycinnamic acid | α-CHC, α-CHCA, 4-HCCA, CHCA | Péptidos, proteínas más pequeñas, triacilgliceroles, otros numerosos compuestos | Ácido 4-cloro-α-ciano-ácido cinámico | ClCCA | Péptidos |
| 3,5-Dimetoxi-4-hidroxicinámico | SA | Proteínas | 2-(4-Hidroxifenilazo) ácido benzoico | HABA | Péptidos, proteínas, glucoproteínas, poliestireno |
| 2-Mercaptobenzotiazol | MBT | Péptidos, proteínas, polímeros sintéticos |
| 5-Cloro-2-mercaptobenzotiazol | CMBT | Glicopéptidos, fosfopéptidos y proteínas |
| 2,6-Dihidroxiacetofenona | DHAP | Glicopéptidos, fosfopéptidos, proteínas |
| 2,4,6-Trihidroxiacetofenona | THAP | Oligonucleótidos de soporte sólido | Ditranol (1,8,9-antracenetriol) | Ninguna | Polímeros sintéticos |
| 9-Nitroantraceno | 9-NA | Fullerenos y derivados | Benzopireno | Nada | Fullerenos y derivados |
| 2-malonitrilo | DCTB | Oligómeros, polímeros, dendrímeros, moléculas pequeñas |
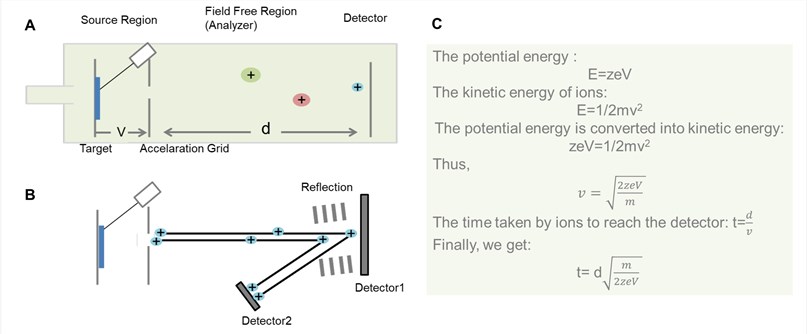
Figura 2. Esquema general del analizador TOF. (A) Analizador TOF de revestimiento; (B) Analizador TOF de reflector; (C) El proceso de derivación del tiempo que los iones pasan por la región libre de campo en el analizador TOF de revestimiento.
- El principio del TOF
Como se muestra en la figura 2, el principio básico del TOF es que los iones de diferentes m/z se dispersan en el tiempo durante su vuelo a lo largo de una trayectoria de deriva libre de campo de longitud conocida. Siempre que todos los iones inicien su viaje al mismo tiempo o, al menos, en un intervalo de tiempo suficientemente corto, los más ligeros llegarán antes al detector que los más pesados.
- Analizador TOF de línea y analizador TOF de reflector
Teóricamente, todos los iones reciben la misma energía cinética inicial, de modo que, tras derivar a lo largo de la región libre de campo, los iones del mismo m/z en el detector en el momento. Sin embargo, en la práctica, el pulso no es percibido por todos los iones con la misma intensidad, por lo que no todos los iones de los mismos valores m/z alcanzan sus velocidades ideales. Para corregir este problema, se suele aplicar un reflector al final de la zona de deriva. El reflector consiste en una serie de electrodos anulares con alto voltaje, que pueden repeler los iones de vuelta a lo largo del tubo de vuelo normalmente en un ángulo ligeramente desplazado.
Los iones de diferente energía cinética penetran en el reflector a diferentes profundidades antes de ser expulsados del reflector en la dirección opuesta. Los iones más rápidos que llevan más energía cinética recorrerán un camino más largo que los más lentos y, por tanto, pasarán más tiempo dentro del reflectrón que los iones más lentos que llevan menos energía. De este modo, el detector recibe iones de la misma masa en (aproximadamente) el mismo tiempo. De este modo, este diseño para el analizador de masas TOF ha aumentado su resolución de forma significativa. Sin embargo, el analizador TOF de reflector no es adecuado para analitos que no son lo suficientemente estables como para sobrevivir al campo eléctrico.
El proceso de espectrometría de masas MALDI-TOF
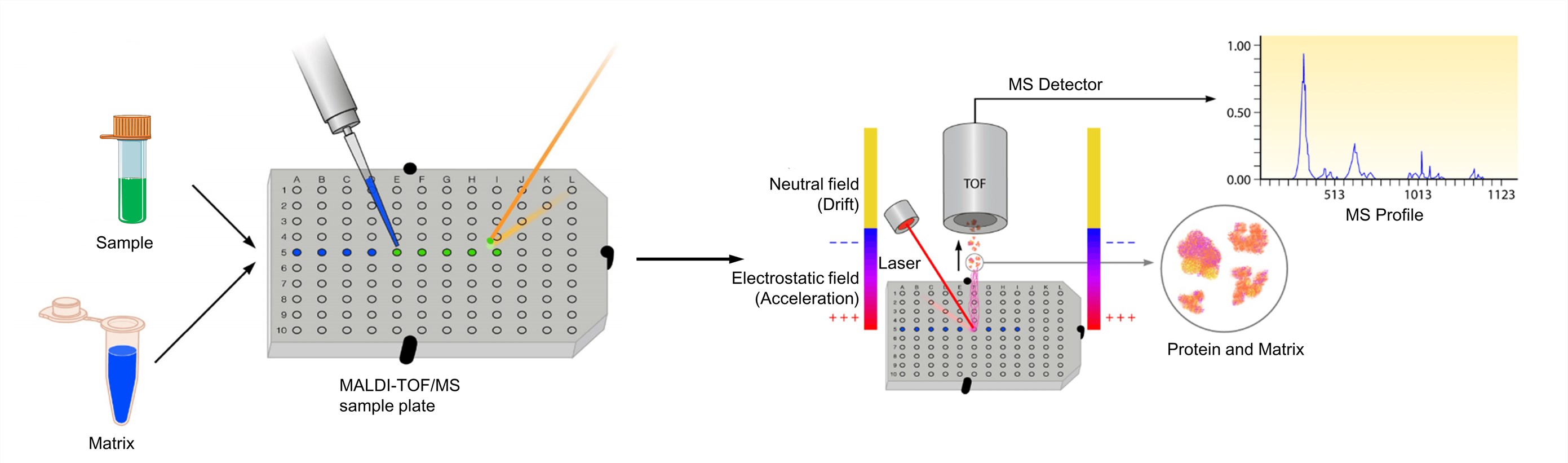
Figura 3. El proceso de la espectrometría de masas MALDI-TOF (Clark A. E., et al.; 2013)
El analito debe ser soluble hasta al menos unos 0,1 mg/ml en algún disolvente. Y la matriz se disuelve para obtener una solución saturada o una concentración de aproximadamente 10 mg/ml. La solución del analito se mezcla entonces con la de la matriz. Para optimizar los espectros MALDI, la relación molar matriz-analito se ajusta normalmente para que caiga en el rango de 1000: 1 a 100.000: 1. A continuación, la mezcla se deposita en una placa metálica para su análisis. Tras el secado, la mezcla de la muestra y la matriz co-cristaliza y forma un depósito sólido de muestra incrustado en la matriz. La placa se carga posteriormente en el instrumento MALDI-TOF y se analiza mediante el software asociado al sistema respectivo. El MALDI conduce a la sublimación e ionización tanto de la muestra como de la matriz. Estos iones generados se separan en función de m/z a través de un analizador TOF, y se genera una representación espectral de estos iones que es analizada por el software de EM, generando un perfil de EM.
Aplicación de la espectrometría de masas MALDI-TOF
- Determinación de la masa intacta
La determinación de la masa intacta es básica e importante para la caracterización de proteínas, debido a que el peso molecular correcto de una proteína puede indicar la estructura intacta. MALDI, una técnica de ionización suave, es adecuada para las proteínas que tienden a ser frágiles y a fragmentarse cuando son ionizadas por otros métodos de ionización. El rendimiento de la EM MALDI-TOF se ve menos afectado por los componentes del tampón, los detergentes y los contaminantes. Además, permite determinar la masa de la proteína intacta con suficiente precisión (≤ 500 ppm) para la validación de la secuencia. Tras la digestión de las proteínas, la EM MALDI-TOF también puede utilizarse para analizar los péptidos obtenidos para la posterior confirmación de la secuencia primaria mediante la huella de masa peptídica.
- Huella de masa peptídica (PMF)
La espectrometría de masas MALDI-TOF tiene un funcionamiento sencillo, una buena precisión de masa, así como una alta resolución y sensibilidad. Por lo tanto, tiene usos generalizados en la proteómica para identificar proteínas a partir de mezclas simples mediante un método denominado huella de masa peptídica, que se utilizan a menudo con la electroforesis en gel bidimensional (2-DE). En este enfoque, los péptidos se generan digiriendo las proteínas de interés con una enzima de secuencia específica como la tripsina. A continuación, los péptidos se analizan mediante espectrometría de masas MALDI-TOF para obtener las masas de los péptidos. Las masas experimentales se comparan con una base de datos que contiene las masas peptídicas teóricas de un organismo determinado con la misma proteasa específica de la secuencia.
- Análisis MALDI-TOF posterior a la descomposición de la fuente (PSD)
Los espectrómetros de masas MALDI-TOF equipados con reflectores pueden analizar iones de fragmentos producidos a partir de iones precursores que se descomponen espontáneamente en el vuelo. Tales iones se denominan generalmente iones metaestables, y el proceso de descomposición en la región libre de campo entre la fuente de iones y el reflector se denomina comúnmente PSD. Los iones de fragmentos PSD se forman dentro de la región libre de campo antes de entrar en el reflector. Los iones de fragmentos PSD pueden separarse, recogerse y registrarse en el detector cambiando continuamente el voltaje del reflector para formar un espectro de masas PSD que proporciona una información estructural muy rica y eficaz para la estructura primaria de péptidos y proteínas. En el estudio proteómico, algunas muestras de proteínas separadas por 2DE no pueden ser identificadas por la PSD o los resultados de la identificación no son claros. La función de secuenciación PSD puede aplicarse a la identificación de estas proteínas. Utilizando la espectroscopia PSD, combinada con una búsqueda en la base de datos, las proteínas pueden ser identificadas rápidamente y con alta especificidad.
- Análisis de oligonucleótidos
Con el desarrollo de las técnicas de biología molecular y las tecnologías de fármacos de ácidos nucleicos antisentido, se han sintetizado cada vez más fragmentos de oligonucleótidos para ser utilizados como cebadores, sondas y fármacos antisentido. Es totalmente necesario detectar rápidamente estos fragmentos para determinar si la síntesis es completa y si la secuencia sintetizada es correcta. La espectrometría de masas, incluida la MALDI-TOF-MS, es con mucho el mejor medio para hacerlo. El análisis de oligonucleótidos mediante MALDI-TOF-MS es sencillo, rápido, preciso y sensible, y puede utilizarse para determinar la secuencia completa del oligonucleótido.
- Imagenización MALDI
El MALDI-TOF puede utilizarse en el perfilado y la imagen de proteínas directamente a partir de secciones finas de tejido, lo que se conoce como espectrometría de masas de imagenización MALDI (MALDI-IMS). Proporciona información específica sobre la composición molecular local, la abundancia relativa y la distribución espacial de péptidos y proteínas en la sección analizada. MALDI-IMS puede analizar múltiples compuestos desconocidos en secciones de tejido biológico simultáneamente a través de una única medición que puede obtener imágenes moleculares del tejido manteniendo la integridad de las células y moléculas en los tejidos.
La espectrometría de masas MALDI-TOF puede analizar una amplia variedad de biomoléculas, como péptidos, proteínas, carbohidratos, oligonucleótidos, etc. Debido al hecho de que los iones formados tienen una baja energía interna, una gran ventaja del MALDI-TOF es que el proceso de ionización suave permite la observación de las moléculas ionizadas con poca a fragmentación de los analitos, lo que permite identificar los iones moleculares de los analitos, incluso dentro de las mezclas. Además, es fácil de usar y mantener, con una rápida adquisición de datos. La elección de la sustancia matriz adecuada es importante para el éxito de la espectrometría de masas MALDI-TOF.
En Creative Proteomics, podemos proporcionar varios servicios basados en nuestras avanzadas plataformas de espectrometría de masas MALDI-TOF, incluyendo:
- Determinación de la masa molecular
- Huellas dactilares de masa de péptidos (PMF)
- Espectrometría de masas por imagen (IMS)
1. Gross J H. Espectrometría de masas: un libro de texto. Springer Science & Business Media, 2006.
2. Boesl U. Time – of – flight mass spectrometry: Introducción a los fundamentos. Mass spectrometry reviews, 2017, 36(1): 86-109.
3. Guerrera I C, Kleiner O. Aplicación de la espectrometría de masas en proteómica. Bioscience Reports, 2005, 25(1-2): 71-93.
4. Fuchs B, Schiller J. Aplicación de la espectrometría de masas MALD – TOF en lipidómica. European Journal of Lipid Science and Technology, 2009, 111(1): 83-98.
5. Duncan M W, Roder H, Hunsucker S W. Quantitative matrix-assisted laser desorption/ionization mass spectrometry. Briefings in functional genomics and proteomics, 2008, 7(5): 355-370.
6. Kenny D J, Brown J M, Palmer M E, et al. A parallel approach to post source decay MALDI-TOF analysis. Journal of the American Society for Mass Spectrometry, 2006, 17(1): 60-66.