Flujo de trabajo WGS: Vista ampliada
Descargar icono pdf
¿Qué es la secuenciación del genoma completo (WGS)?
El genoma, o material genético, de un organismo (bacteria, virus, patata, humano) está formado por ADN. Cada organismo tiene una secuencia de ADN única que está compuesta por bases (A, T, C y G). Si conoce la secuencia de las bases de un organismo, habrá identificado su huella digital o patrón de ADN único. La determinación del orden de las bases se denomina secuenciación. La secuenciación del genoma completo es un procedimiento de laboratorio que determina el orden de las bases en el genoma de un organismo en un solo proceso.
¿Cómo funciona la secuenciación del genoma completo?
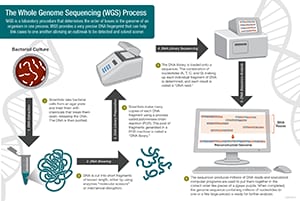
Los científicos llevan a cabo la secuenciación del genoma completo siguiendo estos cuatro pasos principales:
- Corte del ADN: Los científicos comienzan utilizando unas tijeras moleculares para cortar el ADN, que está compuesto por millones de bases: A, C, T y G, en trozos lo suficientemente pequeños para que la máquina de secuenciación pueda leerlos.
- Código de barras de ADN: Los científicos añaden pequeños trozos de etiquetas de ADN, o códigos de barras, para identificar qué trozo de ADN esquilado pertenece a qué bacteria. Esto es similar a la forma en que un código de barras identifica un producto en una tienda de comestibles.
- Secuenciación del genoma completo: El ADN con código de barras de múltiples bacterias se combina y se pone en el secuenciador del genoma completo. El secuenciador identifica las A, C, T y G, o bases, que componen cada secuencia bacteriana. El secuenciador utiliza el código de barras para hacer un seguimiento de qué bases pertenecen a cada bacteria.
- Análisis de datos: Los científicos utilizan herramientas de análisis informático para comparar las secuencias bacterianas e identificar las diferencias. El número de diferencias puede indicar a los científicos lo estrechamente relacionadas que están las bacterias y la probabilidad de que formen parte del mismo brote.

¿Cómo transformará la secuenciación del genoma completo la detección de enfermedades?
La secuenciación del genoma completo proporciona datos más detallados y precisos para identificar los brotes que la técnica estándar actual que utiliza PulseNet, la electroforesis en gel de campo pulsado (PFGE). En lugar de tener sólo la capacidad de comparar genomas bacterianos utilizando 15-30 bandas que aparecen en un patrón de PFGE, ahora tenemos millones de bases para comparar. Es como comparar todas las palabras de un libro (WGS), en lugar de sólo el número de capítulos (PFGE), para ver si los libros son iguales o diferentes. Gracias a la secuenciación del genoma completo, hemos descubierto que algunas bacterias que parecían diferentes mediante PFGE proceden en realidad de la misma fuente. Esto ha ayudado a resolver antes algunos brotes.
La secuenciación del genoma completo es una forma rápida y asequible de obtener información de alto nivel sobre las bacterias utilizando una sola prueba. En la actualidad, el proceso para caracterizar completamente las bacterias requiere que dos o más científicos realicen cuatro o más pruebas separadas, incluida la PFGE. WGS mejorará en gran medida la eficiencia de cómo PulseNet lleva a cabo la vigilancia.
PulseNet está validando activamente la tecnología de secuenciación de próxima generación (NGS), así como desarrollando, evaluando e implementando las herramientas necesarias para analizar los datos.
Cumpliendo con los desafíos de la secuenciación del genoma completo.
En 2013, los CDC comenzaron a utilizar la secuenciación del genoma completo para detectar brotes causados por la mortal bacteria Listeria. Desde entonces, este método ha permitido a los científicos:
- Detectar más grupos de enfermedades por Listeria
- Resolver más brotes de Listeria mientras aún son pequeños
- Enlazar a los pacientes enfermos con las probables fuentes de alimentos
- Identificar nuevas fuentes alimentarias de Listeria, como las manzanas de caramelo y los helados
Aprenda más sobre cómo el Proyecto de Secuenciación del Genoma Completo de la Listeria ha mejorado la detección e investigación de los brotes de origen alimentario.
El CDC está ampliando rápidamente el uso de la secuenciación del genoma completo en los laboratorios estatales, y los científicos pronto comenzarán a utilizar la secuenciación del genoma completo para las investigaciones de brotes de otros patógenos transmitidos por los alimentos, como Campylobacter, E. coli productora de toxina Shiga (STEC) y Salmonella. La iniciativa Advanced Molecular Detection (AMD) de los CDC financió parcialmente la expansión de la WGS en tiempo real para la seguridad alimentaria.
A través de la colaboración con la iniciativa AMD de los CDC y el programa de seguridad alimentaria, PulseNet está estableciendo la estructura para apoyar el cambio a la secuenciación del genoma completo, incluyendo la formación de microbiólogos de salud pública para realizar la secuenciación, la compra de suministros de secuenciación y la actualización de los sistemas para el análisis de datos. Estas actividades son fundamentales para poner en marcha la secuenciación del genoma completo en los laboratorios de salud pública y mejorar la vigilancia de los brotes de enfermedades transmitidas por los alimentos y las tendencias de las infecciones transmitidas por los alimentos y la resistencia a los antibióticos.
A medida que se amplía el uso de la secuenciación del genoma completo, los sistemas nacionales de vigilancia y la infraestructura de los laboratorios de los CDC deben seguir el ritmo de la tecnología cambiante. Con la modernización, los CDC y sus socios de salud pública pueden seguir detectando, respondiendo y deteniendo con éxito las enfermedades infecciosas. Juntos, podemos garantizar diagnósticos rápidos y menos costosos para las personas y las pruebas necesarias para resolver y prevenir rápidamente los brotes de origen alimentario.